非酒精性脂肪性肝炎小鼠模型肝组织T淋巴细胞的特征分析
DOI: 10.12449/JCH250311
Characteristics of T cells in the liver tissues of mice with nonalcoholic steatohepatitis
-
摘要:
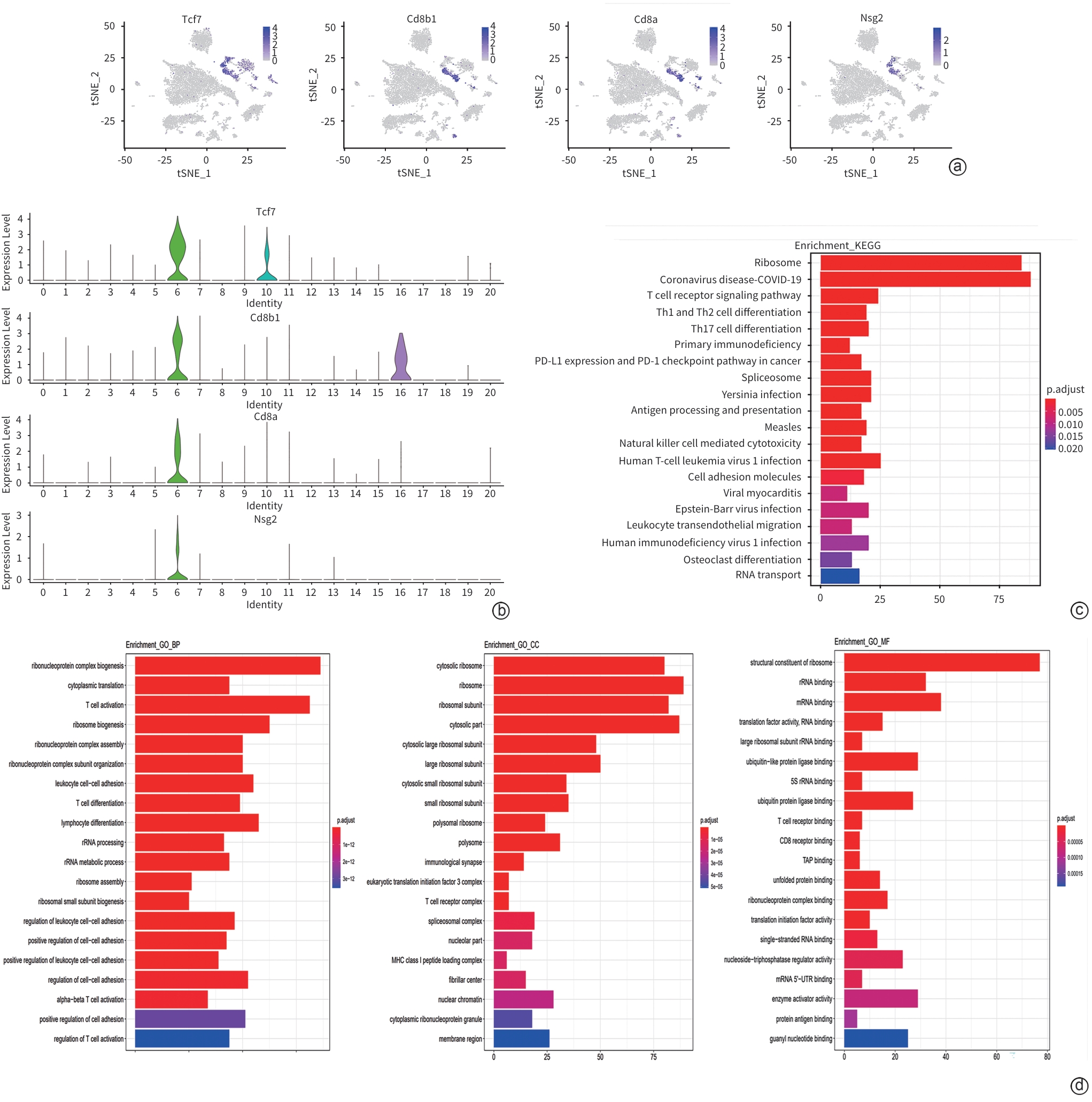
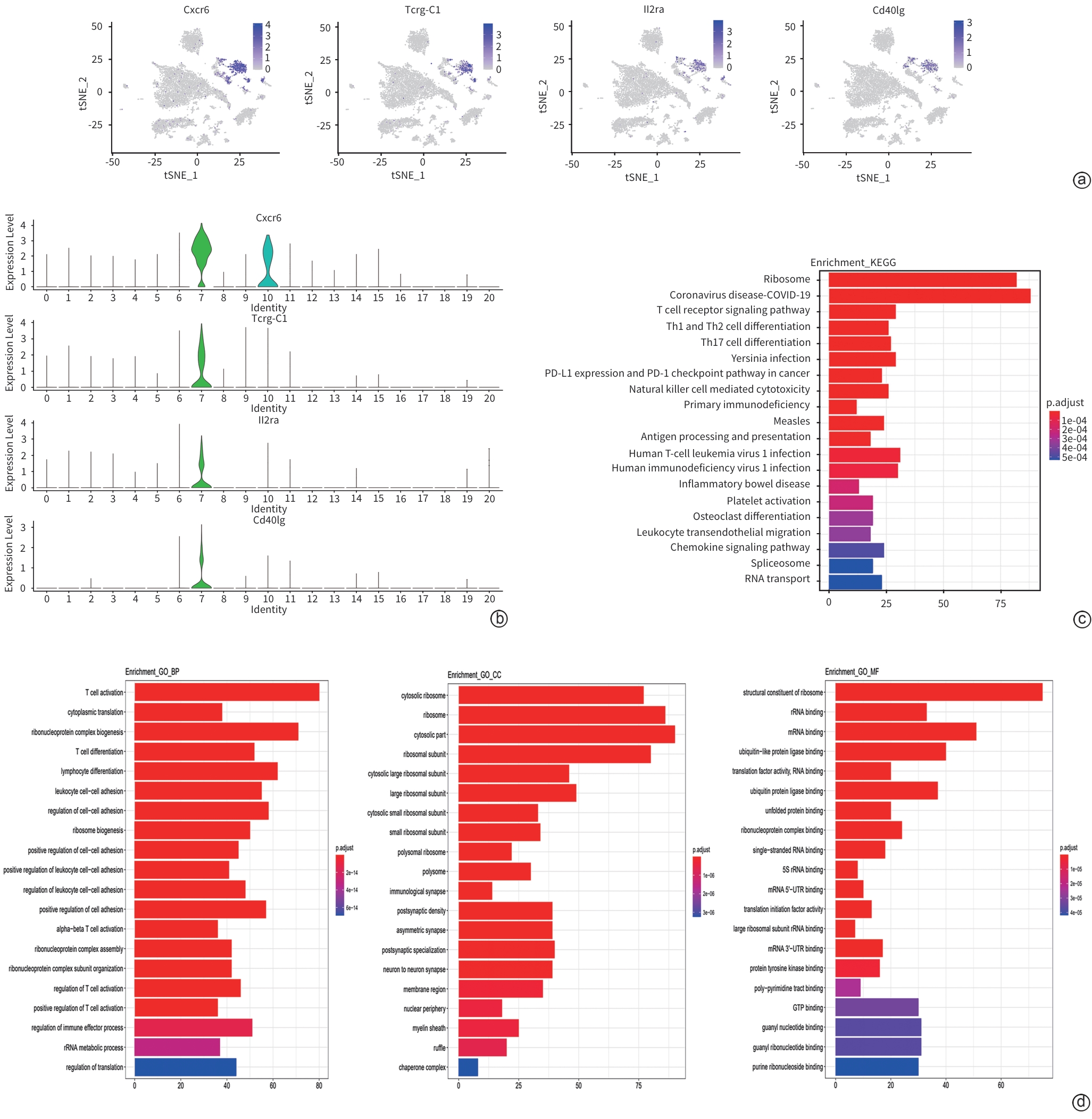
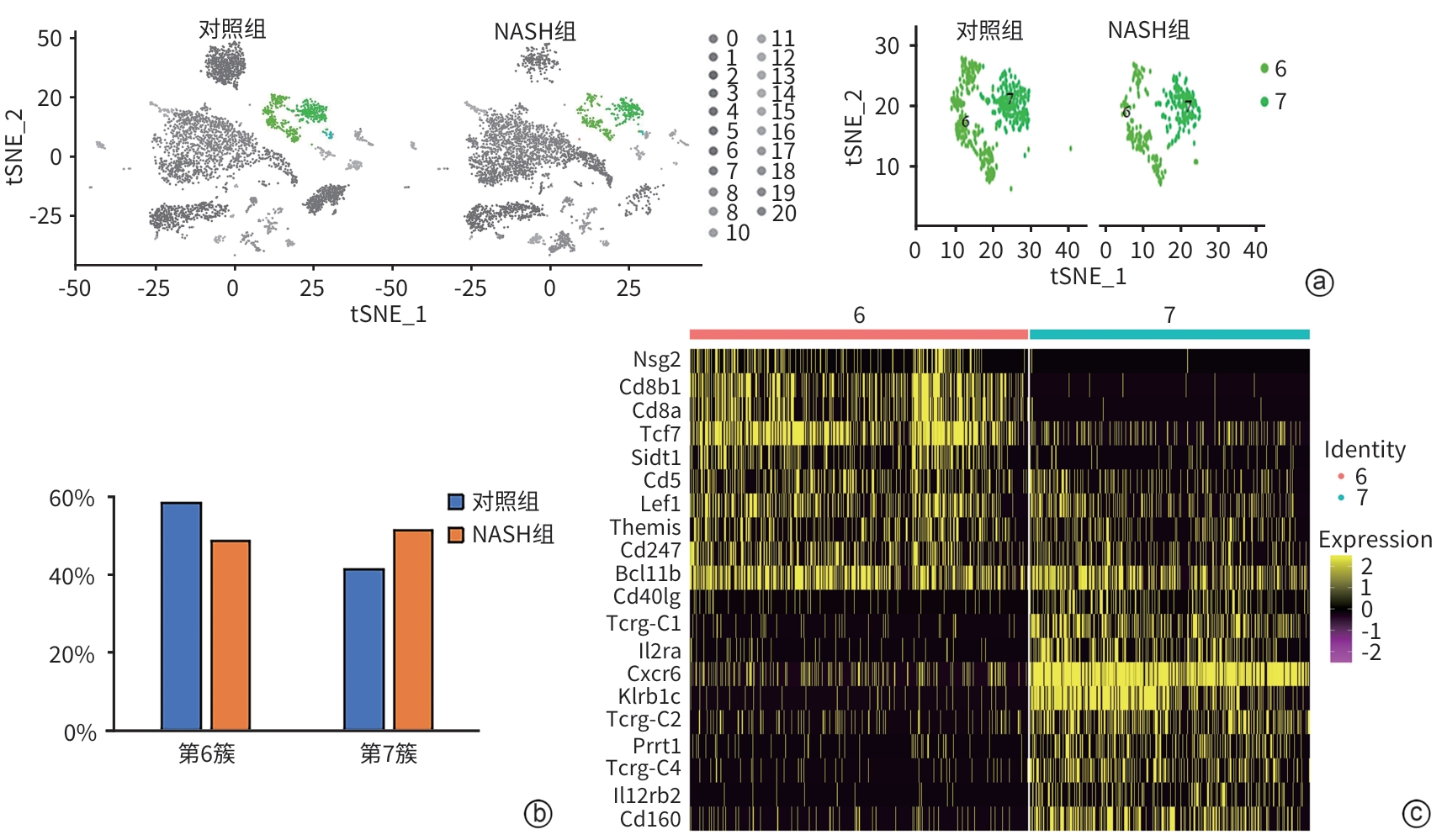
目的 采用单细胞测序技术揭示非酒精性脂肪肝炎(NASH)小鼠肝组织T淋巴细胞单细胞水平的异质性和转录组学特征,为研究T淋巴细胞在NASH中的作用机制提供新的依据。 方法 6只C57BL/6雄性小鼠随机分为普通饲料喂养的对照组和胆碱蛋氨酸缺乏饲料喂养的NASH组,每组各3只。造模6周后取小鼠肝组织进行单细胞RNA测序。分析T淋巴细胞单细胞亚群的特异性差异表达基因,分别行降维聚类、细胞类型注释、t分布随机邻域嵌入(t-SNE)、小提琴图、基因本体(GO)功能富集分析和京都基因与基因组百科全书(KEGG)通路富集分析。利用免疫荧光染色观察两组小鼠肝脏中Tcrα(T淋巴细胞分子标志)、Tcf7、Cxcr6(特征标志基因)的表达情况。计量资料两组间比较采用成组t检验。 结果 小鼠肝脏中共鉴定出2个T淋巴细胞亚群:(1)第6簇T淋巴细胞占比从对照组的58.5%降低到NASH组的48.7%。前4位特异性基因包括Nsg2、Cd8b1、Cd8a和Tcf7。该簇65%的细胞表达Tcf7(第6簇特征标志基因),定义为Tcf7+T淋巴细胞亚群。GO和KEGG富集分析显示,它们参与调控T淋巴细胞活化、白细胞黏附、结合泛素样蛋白连接酶,以及辅助性T淋巴细胞(Th)17、Th1、Th2细胞分化等信号通路。(2)第7簇T淋巴细胞占比从对照组的41.5%增高到NASH组的51.3%。前4位特异性基因包括Cd40lg、Tcrg-C1、Il2rα和Cxcr6。该簇90%的细胞表达Cxcr6,定义为Cxcr6+T淋巴细胞亚群。GO和KEGG富集分析提示,该亚群参与调控T淋巴细胞活化、细胞因子产生,以及T淋巴细胞受体信号通路、Th17细胞分化与MAPK信号通路。免疫荧光结果显示,与对照组相比,NASH组小鼠肝脏中Tcf7蛋白和Tcrα蛋白共染阳性区域减少(1.80%±0.67% vs 0.33%± 0.13%, P<0.05),而Cxcr6蛋白和Tcrα蛋白共染阳性区域增加(0.50%±0.09% vs 2.66%±0.33%, P<0.001)。 结论 NASH小鼠肝脏中Tcf7+T淋巴细胞的占比降低,而Cxcr6+T淋巴细胞的占比增高,揭示了NASH小鼠肝组织T淋巴细胞的特征和差异。 -
关键词:
- 非酒精性脂肪性肝炎 /
- T淋巴细胞 /
- 单细胞测序 /
- 小鼠, 近交C57BL
Abstract:Objective To investigate the heterogeneity and transcriptomic characteristics of T-cell subsets in the liver of mice with nonalcoholic steatohepatitis (NASH) at the single-cell level using single-cell RNA sequencing (scRNA-seq), and to provide a reference for studying the mechanism of action of T cells in NASH. Methods Six male C57BL/6 mice were randomly divided into control group fed with regular diet and NASH group fed with methionine-choline-deficient (MCD) diet, with three mice in each group, and liver tissue was collected for scRNA-seq after 6 weeks of modeling. Specific differentially expressed genes were analyzed between T-cell subsets, and related analyses were performed, including dimension clustering, cell type annotation, t-distributed stochastic neighbor embedding (t-SNE), violin plot, gene ontology (GO) functional enrichment analysis, and Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway enrichment analysis. Immunofluorescent staining was used to observe the expression of the T cell marker Tcrα and the specific marker genes Tcf7 and Cxcr6 in the liver of mice in the two groups. The independent-samples t test was used for comparison of continuous data between two groups. Results Two T cell subsets were identified in the liver of mice, and the percentage of cluster 6 decreased from 58.5% in the control group to 48.7% in the NASH group. The top four specific genes were Nsg2, Cd8b1, Cd8a, and Tcf7. Tcf7, a characteristic marker gene for cluster 6, was expressed in 65% of cells in cluster 6, and therefore, cluster 6 was defined as Tcf7+ T cells. The GO and KEGG enrichment analyses showed that the differentially expressed genes of cluster 6 were involved in T cell activation, leukocyte adhesion, binding ubiquitin-like protein ligase, and the signaling pathways for Th17, Th1, and Th2 cell differentiation. The percentage of cluster 7 increased from 41.5% in the control group to 51.3% in the NASH group. The top four specific genes of cluster 7 were Cd40lg, Tcrg-C1, Il2rα, and Cxcr6. Cxcr6 was expressed in 90% of cells in cluster 7, and therefore, cluster 7 was defined as Cxcr6+ T cells. The GO and KEGG enrichment analyses showed that cluster 7 was involved in T cell activation, cytokine production, the T cell receptor signaling pathway, and the Th17 cell differentiation and MAPK signaling pathway. Immunofluorescence assay showed that compared with the control group, the NASH group showed a significant reduction in the area with positive co-expression of Tcf7 protein and Tcrα protein (1.80%±0.67% vs 0.33%±0.13%, P<0.05) and a significant increase in the area with positive co-expression of Cxcr6 protein and Tcrα protein (0.50%±0.09% vs 2.66%± 0.33%, P<0.001). Conclusion There is a reduction in the percentage of Tcf7+ T cells and an increase in the percentage of Cxcr6+ T cells in NASH mice, revealing the characteristics and differences of T cells in the liver of NASH mice. -
注: DAPI蓝色荧光代表细胞核,Tcrα蛋白红色荧光代表T淋巴细胞。a,绿色荧光为Tcf7,黄色箭头指Tcf7+Tcrα+T淋巴细胞(×630);b,Tcf7+Tcrα+T淋巴细胞占比;c,绿色荧光为Cxcr6,黄色箭头指Cxcr6+Tcrα+T淋巴细胞(×630);d,Cxcr6+Tcrα+T淋巴细胞占比。
图 4 免疫荧光显示T淋巴细胞亚群及特征标志基因的变化
Figure 4. Immunofluorescence shows the changes of T cell subsets and of the expression of the most specific genes between the control and NASH group
表 1 第6簇T细胞前10位特异性基因
Table 1. Top10 specific DEGs of Cluster 6
基因 P值 avg_logFC pct.1 pct.2 校正后的P值 pct_FC Nsg2 5×10-324 0.77 0.25 0.001 5×10-324 253.99 Cd8b1 5×10-324 1.89 0.49 0.012 5×10-324 40.42 Cd8a 5×10-324 1.67 0.44 0.014 5×10-324 31.36 Tcf7 5×10-324 1.95 0.65 0.027 5×10-324 24.22 Sidt1 1.63×10-303 0.92 0.31 0.013 3.89×10-299 24.15 Cd5 4.64×10-292 1.06 0.32 0.016 1.11×10-287 19.99 Lef1 5×10-324 1.24 0.42 0.025 5×10-324 16.64 Themis 3.21×10-241 0.84 0.30 0.018 7.66×10-237 16.44 Cd247 1.03×10-282 0.91 0.35 0.022 2.44×10-278 15.99 Bcl11b 5×10-324 1.57 0.60 0.039 5×10-324 15.44 注:avg_logFC,平均对数倍数变化; pct.1,在第6簇的表达百分比;pct.2,在其他细胞簇的表达百分比; pct-FC,百分比倍数变化。
表 2 第7簇T淋巴细胞前10位特异性基因
Table 2. Top10 specific DEGs of Cluster 7
基因 P值 avg_logFC pct.1 pct.2 校正后的P值 pct_FC Cd40lg 7.69×10-294 0.73 0.26 0.006 1.83×10-289 42.50 Tcrg-C1 5×10-324 1.56 0.46 0.013 5×10-324 35.00 Il2ra 5×10-324 0.99 0.34 0.011 5×10-324 31.00 Cxcr6 5×10-324 2.32 0.90 0.04 5×10-324 22.50 Klrb1c 5×10-324 1.53 0.58 0.03 5×10-324 19.47 Tcrg-C2 5×10-324 1.48 0.44 0.023 5×10-324 19.04 Prrt1 3.39×10-258 0.87 0.32 0.017 8.09×10-254 18.76 Tcrg-C4 7.30×10-290 1.16 0.37 0.02 1.74×10-285 18.50 Il12rb2 9.05×10-193 0.64 0.25 0.014 2.16×10-188 17.93 Cd160 5×10-324 1.12 0.45 0.025 5×10-324 17.80 注:pct.1,在第7簇的表达百分比;pct.2,在其他细胞簇的表达百分比。
-
[1] YOUNOSSI ZM, KOENIG AB, ABDELATIF D, et al. Global epidemiology of nonalcoholic fatty liver disease-Meta-analytic assessment of prevalence, incidence, and outcomes[J]. Hepatology, 2016, 64( 1): 73- 84. DOI: 10.1002/hep.28431. [2] LI J, LI YN, YANG JB, et al. Application of single cell sequencing technology in liver regeneration[J]. Chin J Dig Surg, 2023, 22( 5): 663- 666. DOI: 10.3760/cma.j.cn115610-20230316-00109.李鉴, 李勇男, 杨建宝, 等. 单细胞测序技术在肝再生中的应用[J]. 中华消化外科杂志, 2023, 22( 5): 663- 666. DOI: 10.3760/cma.j.cn115610-20230316-00109. [3] HUNDERTMARK J, BERGER H, TACKE F. Single cell RNA sequencing in NASH[J]. Methods Mol Biol, 2022, 2455: 181- 202. DOI: 10.1007/978-1-0716-2128-8_15. [4] SU Q, KIM SY, ADEWALE F, et al. Single-cell RNA transcriptome landscape of hepatocytes and non-parenchymal cells in healthy and NAFLD mouse liver[J]. iScience, 2021, 24( 11): 103233. DOI: 10.1016/j.isci.2021.103233. [5] KODA Y, TERATANI T, CHU PS, et al. CD8+ tissue-resident memory T cells promote liver fibrosis resolution by inducing apoptosis of hepatic stellate cells[J]. Nat Commun, 2021, 12( 1): 4474. DOI: 10.1038/s41467-021-24734-0. [6] LI WG, ZHOU YW, ZHANG XZ. Level and correlation of peripheral blood hypoxia-inducible factor-1α, regulatory T cell and helper T cell and inflammatory cytokines in patients with non-alcoholic fatty liver disease[J/CD]. Chin J Liver Dis Electron Version, 2020, 12( 4): 83- 88. DOI: 10.3969/j.issn.1674-7380.2020.04.015.李文刚, 周运王, 张学政. 非酒精性脂肪性肝病患者外周血缺氧诱导因子-1α、调节性T细胞和辅助性T细胞17与炎性细胞因子水平及其相关性[J/CD]. 中国肝脏病杂志(电子版), 2020, 12( 4): 83- 88. DOI: 10.3969/j.issn.1674-7380.2020.04.015. [7] LI Y, DU ZX, WANG MT, et al. Correlation between disease progression and levels of T cells in patients with non-alcoholic fatty liver disease[J]. J Clin Med Pract, 2020, 24( 14): 11- 14, 18. DOI: 10.7619/jcmp.202014003.李阳, 杜志祥, 王牧婷, 等. 非酒精性脂肪性肝病患者病情进展与T细胞水平的相关性研究[J]. 实用临床医药杂志, 2020, 24( 14): 11- 14, 18. DOI: 10.7619/jcmp.202014003. [8] LI HY, GAO YX, WU JC, et al. Single-cell transcriptome reveals a novel mechanism of C-Kit+-liver sinusoidal endothelial cells in NASH[J]. Cell Biosci, 2024, 14( 1): 31. DOI: 10.1186/s13578-024-01215-7. [9] National Workshop on Fatty Liver and Alcoholic Liver Disease, Chinese Society of Hepatology, Chinese Medical Association, Fatty Liver Expert Committee, Chinese Medical Doctor Association. Guidelines of prevention and treatment for nonalcoholic fatty liver disease: A 2018 update[J]. J Clin Hepatol, 2018, 34( 5): 947- 957. DOI: 10.3969/j.issn.1001-5256.2018.05.007.中华医学会肝病学分会脂肪肝和酒精性肝病学组, 中国医师协会脂肪性肝病专家委员会. 非酒精性脂肪性肝病防治指南(2018年更新版)[J]. 临床肝胆病杂志, 2018, 34( 5): 947- 957. DOI: 10.3969/j.issn.1001-5256.2018.05.007. [10] YOUNOSSI Z, TACKE F, ARRESE M, et al. Global perspectives on nonalcoholic fatty liver disease and nonalcoholic steatohepatitis[J]. Hepatology, 2019, 69( 6): 2672- 2682. DOI: 10.1002/hep.30251. [11] ZHANG SL, CAO MM, YANG F, et al. Analysis of the change trend of etiological burden of disease of liver cancer in the Chinese population from 1990 to 2019[J]. Chin J Dig Surg, 2023, 22( 1): 122- 130. DOI: 10.3760/cma.j.cn115610-20221112-00687.张绍丽, 曹毛毛, 杨帆, 等. 1990—2019年中国人群肝癌各病因疾病负担变化趋势分析[J]. 中华消化外科杂志, 2023, 22( 1): 122- 130. DOI: 10.3760/cma.j.cn115610-20221112-00687. [12] HARDY T, OAKLEY F, ANSTEE QM, et al. Nonalcoholic fatty liver disease: Pathogenesis and disease spectrum[J]. Annu Rev Pathol, 2016, 11: 451- 496. DOI: 10.1146/annurev-pathol-012615-044224. [13] NOUREDDIN M, SANYAL AJ. Pathogenesis of NASH: The impact of multiple pathways[J]. Curr Hepatol Rep, 2018, 17( 4): 350- 360. DOI: 10.1007/s11901-018-0425-7. [14] WOLF MJ, ADILI A, PIOTROWITZ K, et al. Metabolic activation of intrahepatic CD8+ T cells and NKT cells causes nonalcoholic steatohepatitis and liver cancer via cross-talk with hepatocytes[J]. Cancer Cell, 2014, 26( 4): 549- 564. DOI: 10.1016/j.ccell.2014.09.003. [15] HER Z, TAN JHL, LIM YS, et al. CD4+ T cells mediate the development of liver fibrosis in high fat diet-induced NAFLD in humanized mice[J]. Front Immunol, 2020, 11: 580968. DOI: 10.3389/fimmu.2020.580968. [16] ZHANG YZ, FU BQ, NIU CY, et al. The development of single cell sequencing[J]. Acta Metrol Sin, 2023, 44( 1): 149- 156. DOI: 10.3969/j.issn.1000-1158.2023.01.22.张永卓, 傅博强, 牛春艳, 等. 单细胞测序技术的发展[J]. 计量学报, 2023, 44( 1): 149- 156. DOI: 10.3969/j.issn.1000-1158.2023.01.22. [17] LI DR, ZHANG ZJ, ZHANG C, et al. Unraveling the connection between Hashimoto’s Thyroiditis and non-alcoholic fatty liver disease: Exploring the role of CD4+ central memory T cells through integrated genetic approaches[J]. Endocrine, 2024, 85( 2): 751- 765. DOI: 10.1007/s12020-024-03745-z. [18] HUANG YY, LIU X, WANG HY, et al. Single-cell transcriptome landscape of zebrafish liver reveals hepatocytes and immune cell interactions in understanding nonalcoholic fatty liver disease[J]. Fish Shellfish Immunol, 2024, 146: 109428. DOI: 10.1016/j.fsi.2024.109428. [19] YU Q, SHARMA A, OH SY, et al. T cell factor 1 initiates the T helper type 2 fate by inducing the transcription factor GATA-3 and repressing interferon-gamma[J]. Nat Immunol, 2009, 10( 9): 992- 999. DOI: 10.1038/ni.1762. [20] ROLLA S, ALCHERA E, IMARISIO C, et al. The balance between IL-17 and IL-22 produced by liver-infiltrating T-helper cells critically controls NASH development in mice[J]. Clin Sci(Lond), 2016, 130( 3): 193- 203. DOI: 10.1042/CS20150405. [21] WANG ZY. Review on the mechanism of T-cell factor 3 in the progression and metastasis of hepatocellular carcinoma[J]. J World Latest Med Inf, 2024, 24( 16): 43- 46. DOI: 10.3969/j.issn.1671-3141.2024.016.009.王梓伊. T细胞因子3促进肝癌侵袭和转移的机制的综述[J]. 世界最新医学信息文摘, 2024, 24( 16): 43- 46. DOI: 10.3969/j.issn.1671-3141.2024.016.009. [22] GUI MH, HUANG SL, LI SZ, et al. Integrative single-cell transcriptomic analyses reveal the cellular ontological and functional heterogeneities of primary and metastatic liver tumors[J]. J Transl Med, 2024, 22( 1): 206. DOI: 10.1186/s12967-024-04947-9. [23] UNUTMAZ D, XIANG W, SUNSHINE MJ, et al. The primate lentiviral receptor Bonzo/STRL33 is coordinately regulated with CCR5 and its expression pattern is conserved between human and mouse[J]. J Immunol, 2000, 165( 6): 3284- 3292. DOI: 10.4049/jimmunol.165.6.3284. [24] DUDEK M, PFISTER D, DONAKONDA S, et al. Auto-aggressive CXCR6+ CD8 T cells cause liver immune pathology in NASH[J]. Nature, 2021, 592( 7854): 444- 449. DOI: 10.1038/s41586-021-03233-8. [25] WEHR A, BAECK C, HEYMANN F, et al. Chemokine receptor CXCR6-dependent hepatic NK T Cell accumulation promotes inflammation and liver fibrosis[J]. J Immunol, 2013, 190( 10): 5226- 5236. DOI: 10.4049/jimmunol.1202909. -


 PDF下载 ( 3813 KB)
PDF下载 ( 3813 KB)

 下载:
下载: