基于生物信息学分析Midkine在胆管癌中的表达及其对预后的预测价值
DOI: 10.12449/JCH240722
Expression of Midkine in cholangiocarcinoma and its value in predicting prognosis based on bioinformatics analysis
-
摘要:
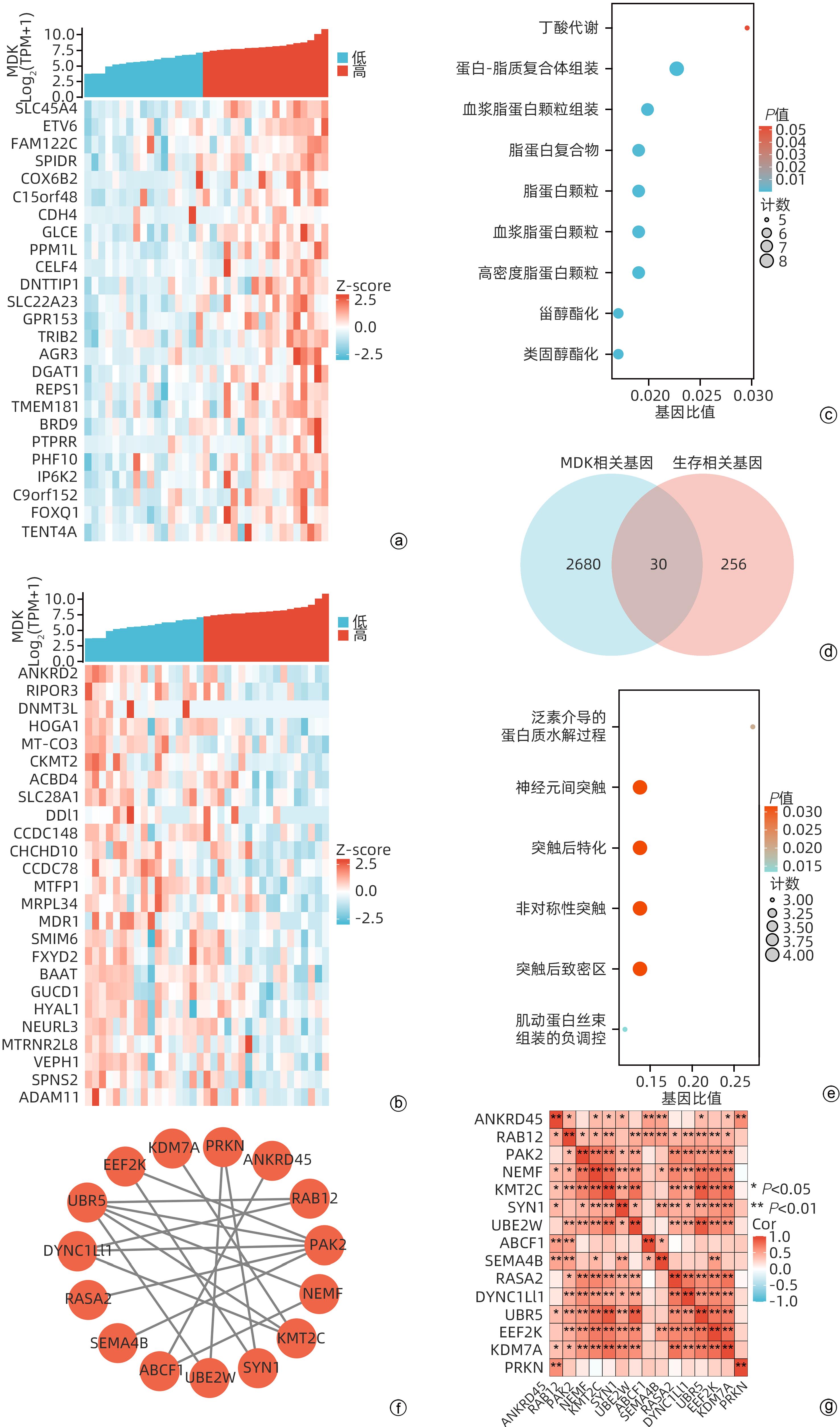
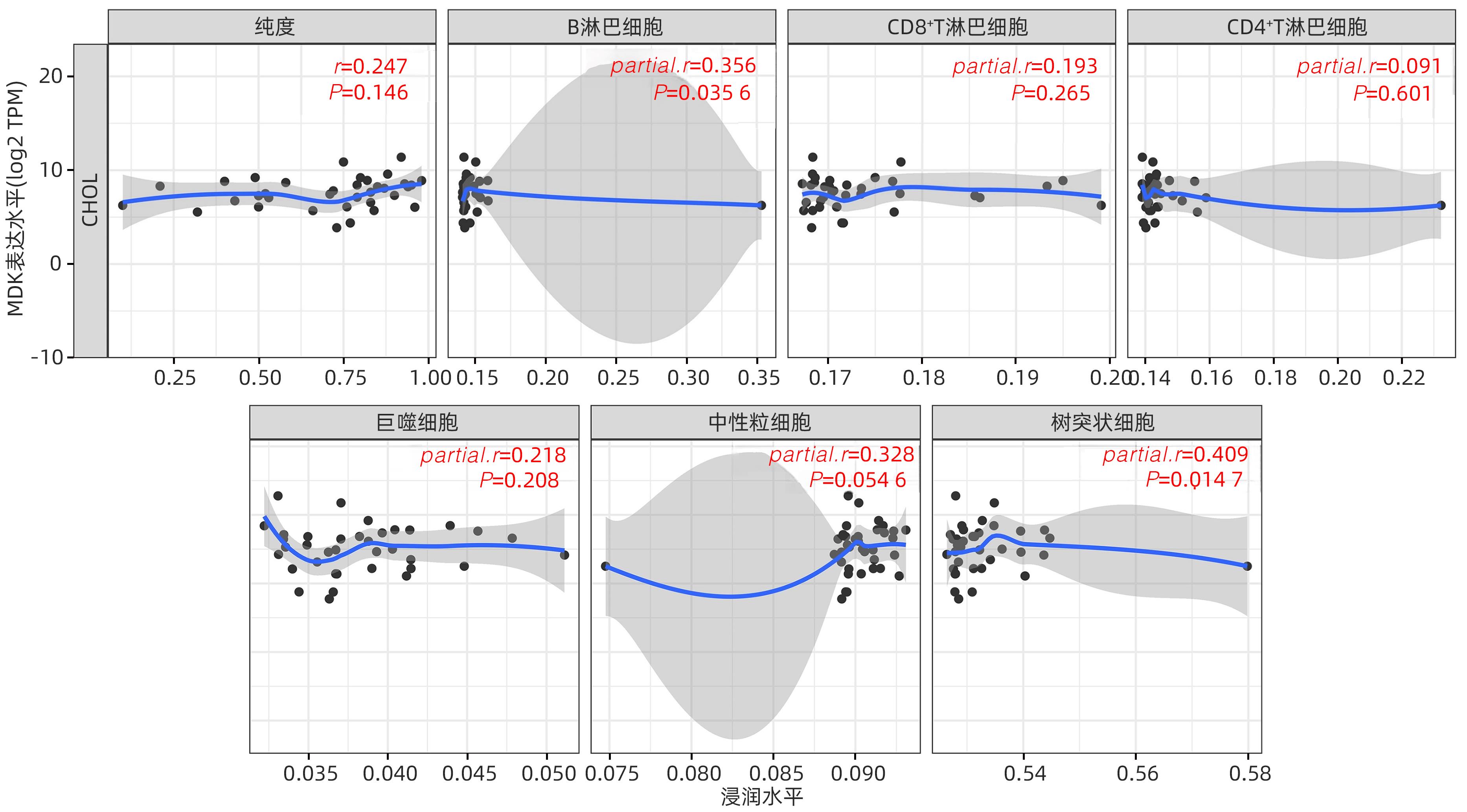
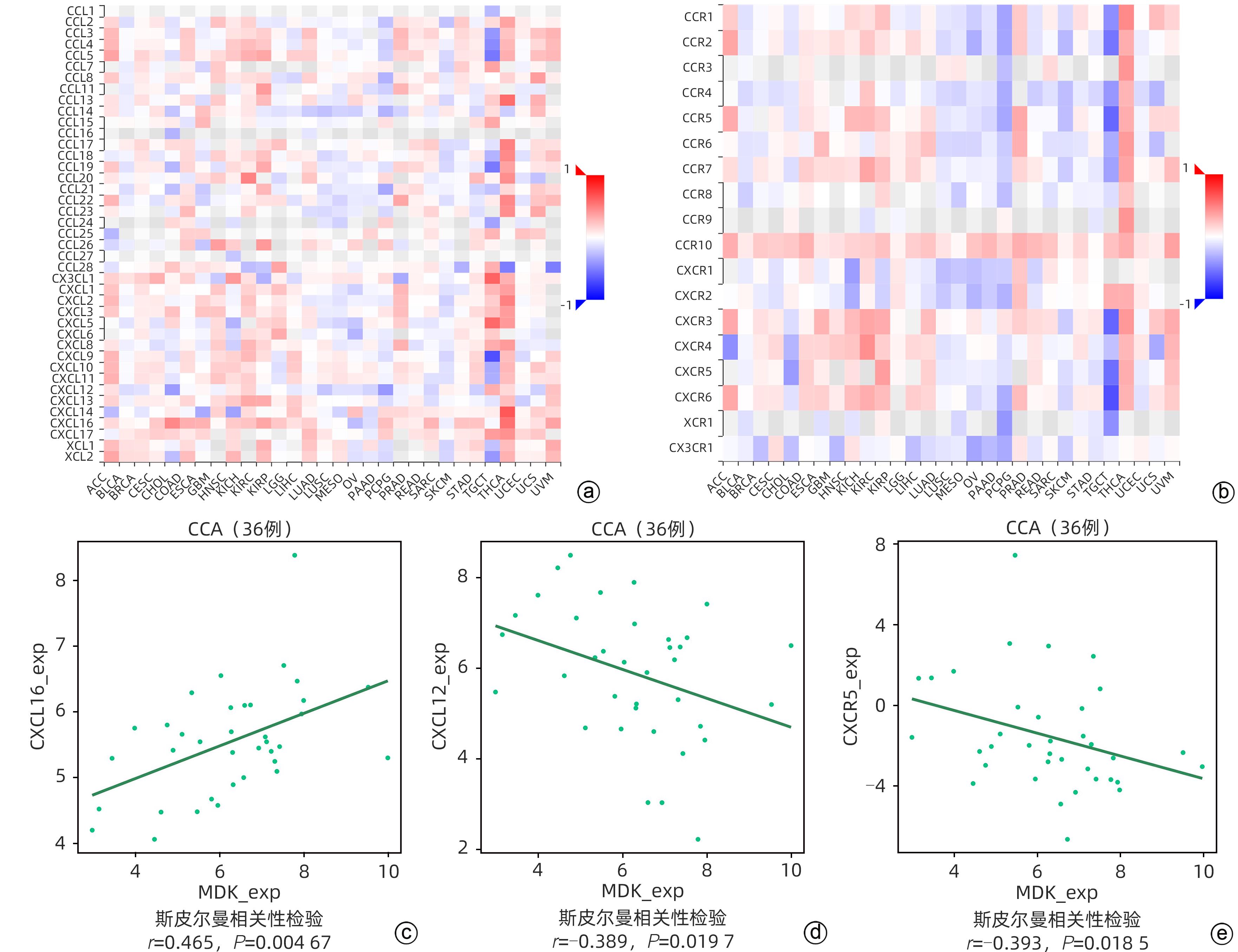
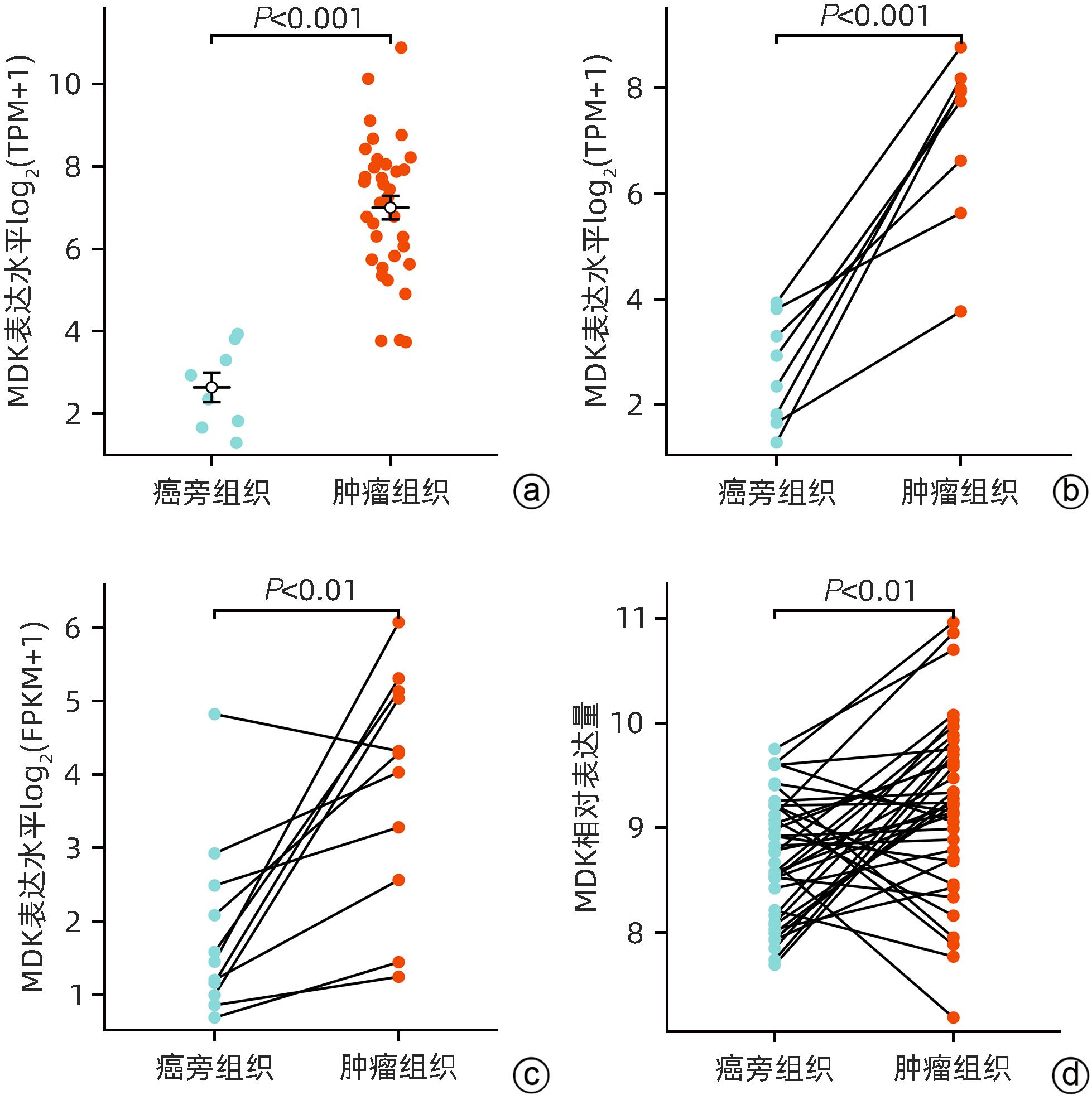
目的 研究中期因子(MDK)在胆管癌(CCA)中的表达及其对患者预后的预测价值,探索MDK可能影响CCA进展的机制。 方法 从癌症基因组图谱(TCGA)数据库获取CCA样本数据信息,分析MDK在癌与癌旁组织中的表达差异及其与临床特征的相关性,使用基因表达综合数据库(GEO)和解放军总医院第五医学中心2018年6月—2021年9月经手术切除的11例CCA患者的临床资料进行相关验证。通过STRING和Cytoscape构建蛋白互作网络,使用基因本体(GO)和京都基因与基因组数据库(KEGG)进行富集分析,探讨MDK相关基因所参与的生物学过程和肿瘤相关通路。此外,应用TIMER和TISIDB数据库分析CCA肿瘤组织中MDK的表达与免疫细胞浸润的相关性。计量资料两组间比较采用成组t检验或Mann-Whitney U检验。计数资料组间比较采用Fisher精确概率法。应用Kaplan-Meier法绘制生存曲线,并采用Log-rank检验进行组间比较。两变量间的相关性采用Spearman相关性分析。 结果 比较TCGA数据库中CCA患者肿瘤组织及癌旁组织中MDK的表达水平,非配对分析结果和配对分析结果均显示,MDK在CCA肿瘤组织中的表达水平高于癌旁组织(P值均<0.001);对纳入的11例CCA患者肿瘤组织及其配对癌旁组织标本进行转录组测序,结果显示,MDK在CCA肿瘤组织中的表达水平显著高于其配对癌旁组织(P<0.01)。MDK高表达与淋巴结转移(P=0.045)和血管侵犯(P=0.044)有关。生存分析显示,MDK高表达的CCA患者总生存期(χ2=5.30,P=0.028)及疾病特异性生存期(χ2=6.25,P=0.019)均明显短于MDK低表达的患者。GO和KEGG富集分析显示,MDK表达相关的30个基因与泛素介导的蛋白质水解过程密切相关,并影响CCA患者预后;TIMER分析结果显示,MDK表达与B淋巴细胞(r=0.356,P=0.035 6)和树突状细胞(r=0.409,P=0.014 7)在肿瘤组织微环境中的浸润呈正相关;TISIDB分析结果显示,MDK表达水平与CXCL16(r=0.465,P=0.004 67)呈正相关,与CXCL12(r=-0.389,P=0.019 7)及CXCR5(r=-0.393,P=0.018 5)呈负相关;与免疫检查点调节剂VTCN1(r=-0.393,P=0.018 3)、LTA(r=-0.380,P=0.022 7)和PVR(r=-0.350,P=0.037 3)呈负相关。 结论 MDK高表达提示CCA患者预后不佳,MDK有潜力成为预测CCA患者预后的分子标志物,其可能通过调控泛素介导的蛋白质水解过程及调控B淋巴细胞和树突状细胞的浸润促进CCA的发生发展。 Abstract:Objective To investigate the expression of Midkine (MDK) in cholangiocarcinoma (CCA) and its value in predicting the prognosis of CCA, as well as the potential mechanism of the effect of MDK on the progression of CCA. Methods The data of CCA samples were obtained from TCGA database to analyze the difference in the expression of MDK between cancer tissue and paracancerous tissue and its association with clinical features, and the data collected from GEO database and 11 CCA patients who underwent surgical resection in The Fifth Medical Center of Chinese PLA General Hospital from June 2018 to September 2021 were used for validation. STRING and Cytoscape were used to construct a protein-protein interaction network, and gene ontology (GO) and Kyoto Encyclopedia of Genes and Genomes (KEGG) enrichment analyses were used to investigate the biological functions and tumor-related pathways involving MDK-related genes. In addition, TIMER and TISIDB databases were used to analyze the correlation between MDK expression and immune cell infiltration in CCA tissue. The independent-samples t test or the Mann-Whitney U test was used for comparison of continuous data between two groups, and the Fisher’s exact test was used for comparison of categorical data between two groups. The Kaplan-Meier method was used to plot survival curves, and the Log-rank test was used for comparison between groups. The Spearman correlation analysis was used to investigate the correlation between two variables. Results The expression level of MDK in cancer tissue and paracancerous tissue of CCA patients was compared based on TCGA database, and the results of the non-paired and paired analyses showed that the expression level of MDK in CCA tumor tissue was significantly higher than that in paracancerous tissue (P<0.001). Transcriptome sequencing was performed for the tumor tissue and its corresponding paracancerous tissue from 11 CCA patients, and the results showed that the expression level of MDK in CCA tumor tissue was significantly higher than that in corresponding paracancerous tissue (P<0.01). High expression of MDK was associated with lymph node metastasis (P=0.045) and vascular invasion (P=0.044). Survival analysis showed that compared with the CCA patients with low MDK expression, the CCA patients with high MDK expression had significantly shorter overall survival time (χ2=5.30, P=0.028) and disease-specific survival time (χ2=6.25, P=0.019). The GO and KEGG enrichment analyses showed that the 30 MDK-related genes were closely associated with ubiquitin-mediated proteolysis and affected the prognosis of CCA patients. The TIMER analysis showed that the expression level of MDK was positively correlated with the infiltration of B cells (r=0.356, P=0.035 6) and dendritic cells (r=0.409, P=0.014 7) in tumor microenvironment of CCA; the TISIDB analysis showed that the expression level of MDK was positively correlated with CXCL16 (r=0.465, P=0.004 67) and was negatively correlated with CXCL12 (r=-0.389, P=0.019 7) and CXCR5 (r=-0.393, P=0.018 5), and it was also negatively correlated with the immune checkpoint regulators VTCN1 (r=-0.393, P=0.018 3), LTA (r=-0.380, P=0.022 7), and PVR (r=-0.350, P=0.037 3). Conclusion High expression of MDK is associated with poor prognosis in CCA patients, and MDK has the potential of being used as a molecular marker for predicting the prognosis of CCA. MDK may promote the development and progression of CCA by regulating ubiquitin-mediated proteolysis and the infiltration of B cells and dendritic cells. -
Key words:
- Cholangiocarcinoma /
- Midkine /
- Prognosis
-
表 1 TCGA数据库中MDK表达与CCA患者临床特征的关系
Table 1. The relationship of MDK expression and clinical characteristics in patients with cholangiocarcinoma from TCGA
临床指标 低表达(n=17) 高表达(n=18) P值 T分期[例(%)] >0.05 T1期 9(52.9) 9(50.0) T2期 6(35.3) 6(33.3) T3/T4期 2(11.8) 3(16.7) 淋巴结转移[例(%)]1) 0.045 否 14(100.0) 11(68.8) 是 0(0.0) 5(31.3) M分期[例(%)]2) >0.05 M0期 13(86.7) 14(82.4) M1期 2(13.3) 3(17.6) 性别[例(%)] >0.05 女 9(52.9) 10(55.6) 男 8(47.1) 8(44.4) 年龄[例(%)] 0.318 ≤65岁 10(58.8) 7(38.9) >65岁 7(41.2) 11(61.1) BMI[例(%)]3) >0.05 ≤25 kg/m2 5(31.3) 5(27.8) >25 kg/m2 11(68.8) 13(72.2) 血管侵犯[例(%)]4) 0.044 否 16(100.0) 12(70.6) 是 0(0.0) 5(29.4) 注:1) MDK低表达组3例患者数据缺失,高表达组2例患者数据缺失;2) MDK低表达组2例患者数据缺失,高表达组1例患者数据缺失;3) MDK低表达组1例患者数据缺失;4) MDK低表达组和高表达组各有1例患者数据缺失。 表 2 临床收集的11例CCA患者肿瘤组织中MDK的表达与临床特征参数的关系
Table 2. The relationship of MDK expression and clinical characteristics in 11 patients with cholangiocarcinoma
临床指标 例数 MDK表达水平 t值 P值 年龄 0.567 0.586 ≤60岁 7 3.71±1.95 >60岁 4 4.18±0.77 性别 1.487 0.171 女 5 4.62±1.05 男 6 3.27±1.78 BMI 2.543 0.075 ≤25 kg/m2 7 3.15±1.47 >25 kg/m2 4 5.17±0.74 肿瘤直径 0.538 0.601 ≤5 cm 3 3.39±0.88 >5 cm 8 4.07±1.80 肿瘤数目 1.146 0.282 单发 3 3.00±1.44 多发 8 4.22±1.59 淋巴结转移 2.416 0.039 否 3 2.34±1.72 是 8 4.47±1.15 血管侵犯 0.578 0.577 否 9 4.02±1.41 是 2 3.28±2.88 表 3 TCGA数据库中CCA患者的单因素Cox回归分析
Table 3. Univariate Cox regression analyses of cholangiocarcinoma from TCGA
参数 例数 HR(95%CI) P值 T分期 T1/T2期 30 1.000 T3/T4期 5 0.615(0.140~2.700) 0.520 淋巴结转移1) 否 25 1.000 是 5 2.147(0.565~8.154) 0.262 M分期2) M0期 27 1.000 M1期 5 1.531(0.428~5.475) 0.513 年龄 ≤65岁 17 1.000 >65岁 18 1.461(0.570~3.747) 0.430 性别 女 19 1.000 男 16 1.279(0.503~3.255) 0.605 BMI3) ≤25 kg/m2 10 1.000 >25 kg/m2 24 0.552(0.200~1.527) 0.253 血管侵犯4) 否 28 1.000 是 5 1.665(0.461~6.012) 0.436 神经侵犯5) 否 25 1.000 是 7 4.056(1.128~14.582) 0.032 MDK 低表达 17 1.000 高表达 18 3.374(1.141~9.976) 0.028 注:1) 5例患者淋巴结转移数据缺失;2) 3例患者M分期数据缺失;3) 1例患者BMI数据缺失;4) 2例患者血管侵犯数据缺失;5) 3例患者神经侵犯数据缺失。 -
[1] GRETEN TF, SCHWABE R, BARDEESY N, et al. Immunology and immunotherapy of cholangiocarcinoma[J]. Nat Rev Gastroenterol Hepatol, 2023, 20( 6): 349- 365. DOI: 10.1038/s41575-022-00741-4. [2] BANALES JM, MARIN JJG, LAMARCA A, et al. Cholangiocarcinoma 2020: The next horizon in mechanisms and management[J]. Nat Rev Gastroenterol Hepatol, 2020, 17( 9): 557- 588. DOI: 10.1038/s41575-020-0310-z. [3] VALLE JW, KELLEY RK, NERVI B, et al. Biliary tract cancer[J]. Lancet, 2021, 397( 10272): 428- 444. DOI: 10.1016/S0140-6736(21)00153-7. [4] NAGINO M, HIRANO S, YOSHITOMI H, et al. Clinical practice guidelines for the management of biliary tract cancers 2019: The 3rd English edition[J]. J Hepatobiliary Pancreat Sci, 2021, 28( 1): 26- 54. DOI: 10.1002/jhbp.870. [5] LI Y, LI DJ, CHEN J, et al. Application of joint detection of AFP, CA19-9, CA125 and CEA in identification and diagnosis of cholangiocarcinoma[J]. Asian Pac J Cancer Prev, 2015, 16( 8): 3451- 3455. DOI: 10.7314/apjcp.2015.16.8.3451. [6] RAZUMILAVA N, GORES GJ. Cholangiocarcinoma[J]. Lancet, 2014, 383( 9935): 2168- 2179. DOI: 10.1016/S0140-6736(13)61903-0. [7] ZHANG ZZ, WANG G, YIN SH, et al. Midkine: A multifaceted driver of atherosclerosis[J]. Clin Chim Acta, 2021, 521: 251- 257. DOI: 10.1016/j.cca.2021.07.024. [8] CAMPBELL VK, GATELY RP, KRISHNASAMY R, et al. Midkine and chronic kidney disease-associated multisystem organ dysfunctions[J]. Nephrol Dial Transplant, 2021, 36( 9): 1577- 1584. DOI: 10.1093/ndt/gfaa084. [9] FILIPPOU PS, KARAGIANNIS GS, CONSTANTINIDOU A. Midkine(MDK) growth factor: A key player in cancer progression and a promising therapeutic target[J]. Oncogene, 2020, 39( 10): 2040- 2054. DOI: 10.1038/s41388-019-1124-8. [10] CHOI YW, KIM YH, LEE J, et al. Strong immunoexpression of midkine is associated with multiple lymph node metastases in BRAFV600E papillary thyroid carcinoma[J]. Hum Pathol, 2015, 46( 10): 1557- 1565. DOI: 10.1016/j.humpath.2015.06.018. [11] GÜNGÖR C, ZANDER H, EFFENBERGER KE, et al. Notch signaling activated by replication stress-induced expression of midkine drives epithelial-mesenchymal transition and chemoresistance in pancreatic cancer[J]. Cancer Res, 2011, 71( 14): 5009- 5019. DOI: 10.1158/0008-5472.CAN-11-0036. [12] YAO X, WANG X, WANG ZS, et al. Clinicopathological and prognostic significance of epithelial mesenchymal transition-related protein expression in intrahepatic cholangiocarcinoma[J]. Onco Targets Ther, 2012, 5: 255- 261. DOI: 10.2147/OTT.S36213. [13] ZHANG YJ, ZUO CM, LIU LG, et al. Single-cell RNA-sequencing atlas reveals an MDK-dependent immunosuppressive environment in ErbB pathway-mutated gallbladder cancer[J]. J Hepatol, 2021, 75( 5): 1128- 1141. DOI: 10.1016/j.jhep.2021.06.023. [14] WANG D, BU F, ZHANG WW. The role of ubiquitination in regulating embryonic stem cell maintenance and cancer development[J]. Int J Mol Sci, 2019, 20( 11): 2667. DOI: 10.3390/ijms20112667. [15] CHEN Y, XU X, WANG YR, et al. Hypoxia-induced SKA3 promoted cholangiocarcinoma progression and chemoresistance by enhancing fatty acid synthesis via the regulation of PAR-dependent HIF-1a deubiquitylation[J]. J Exp Clin Cancer Res, 2023, 42( 1): 265. DOI: 10.1186/s13046-023-02842-7. [16] CEREZO-WALLIS D, CONTRERAS-ALCALDE M, TROULÉ K, et al. Midkine rewires the melanoma microenvironment toward a tolerogenic and immune-resistant state[J]. Nat Med, 2020, 26( 12): 1865- 1877. DOI: 10.1038/s41591-020-1073-3. [17] ZHAO SL, WANG HJ, NIE YZ, et al. Midkine upregulates MICA/B expression in human gastric cancer cells and decreases natural killer cell cytotoxicity[J]. Cancer Immunol Immunother, 2012, 61( 10): 1745- 1753. DOI: 10.1007/s00262-012-1235-3. [18] GUO XF, PAN Y, XIONG M, et al. Midkine activation of CD8+ T cells establishes a neuron-immune-cancer axis responsible for low-grade glioma growth[J]. Nat Commun, 2020, 11( 1): 2177. DOI: 10.1038/s41467-020-15770-3. [19] FLORES-BORJA F, BLAIR P. Mechanisms of induction of regulatory B cells in the tumour microenvironment and their contribution to immunosuppression and pro-tumour responses[J]. Clin Exp Immunol, 2022, 209( 1): 33- 45. DOI: 10.1093/cei/uxac029. [20] SHANG J, ZHA HR, SUN YF. Phenotypes, functions, and clinical relevance of regulatory B cells in cancer[J]. Front Immunol, 2020, 11: 582657. DOI: 10.3389/fimmu.2020.582657. [21] MICHAUD D, STEWARD CR, MIRLEKAR B, et al. Regulatory B cells in cancer[J]. Immunol Rev, 2021, 299( 1): 74- 92. DOI: 10.1111/imr.12939. [22] KATOPODI T, PETANIDIS S, CHARALAMPIDIS C, et al. Tumor-infiltrating dendritic cells: Decisive roles in cancer immunosurveillance, immunoediting, and tumor T cell tolerance[J]. Cells, 2022, 11( 20): 3183. DOI: 10.3390/cells11203183. [23] MOLLICA POETA V, MASSARA M, CAPUCETTI A, et al. Chemokines and chemokine receptors: New targets for cancer immunotherapy[J]. Front Immunol, 2019, 10: 379. DOI: 10.3389/fimmu.2019.00379. [24] KORBECKI J, BAJDAK-RUSINEK K, KUPNICKA P, et al. The role of CXCL16 in the pathogenesis of cancer and other diseases[J]. Int J Mol Sci, 2021, 22( 7): 3490. DOI: 10.3390/ijms22073490. [25] MEZZAPELLE R, LEO M, CAPRIOGLIO F, et al. CXCR4/CXCL12 activities in the tumor microenvironment and implications for tumor immunotherapy[J]. Cancers, 2022, 14( 9): 2314. DOI: 10.3390/cancers14092314. [26] SILIŅA K, SOLTERMANN A, ATTAR FM, et al. Germinal centers determine the prognostic relevance of tertiary lymphoid structures and are impaired by corticosteroids in lung squamous cell carcinoma[J]. Cancer Res, 2018, 78( 5): 1308- 1320. DOI: 10.1158/0008-5472.CAN-17-1987. [27] XIE N, CAI JB, ZHANG L, et al. Upregulation of B7-H4 promotes tumor progression of intrahepatic cholangiocarcinoma[J]. Cell Death Dis, 2017, 8( 12): 3205. DOI: 10.1038/s41419-017-0015-6. [28] KAMIYA T, OHTANI N. The role of immune cells in the liver tumor microenvironment: An involvement of gut microbiota-derived factors[J]. Int Immunol, 2022, 34( 9): 467- 474. DOI: 10.1093/intimm/dxac020. [29] QU P, HUANG XJ, ZHOU XC, et al. Loss of CD155 expression predicts poor prognosis in hepatocellular carcinoma[J]. Histopathology, 2015, 66( 5): 706- 714. DOI: 10.1111/his.12584. [30] DU XN, ALMEIDA PD, MANIERI N, et al. CD226 regulates natural killer cell antitumor responses via phosphorylation-mediated inactivation of transcription factor FOXO1[J]. Proc Natl Acad Sci U S A, 2018, 115( 50): E11731- E11740. DOI: 10.1073/pnas.1814052115. -


 PDF下载 ( 2483 KB)
PDF下载 ( 2483 KB)

 下载:
下载: