固醇调节元件结合蛋白(SREBP)在非酒精性脂肪性肝病中的作用机制及治疗靶点
DOI: 10.12449/JCH240726
Mechanism of action of sterol regulatory element-binding proteins in nonalcoholic fatty liver disease and related therapeutic targets
-
摘要: 非酒精性脂肪性肝病(NAFLD)已成为全球范围内最常见的肝脏疾病,是肝癌发展的重要风险因素。然而,NAFLD的发病机制目前仍未被完全阐明,且尚无特异性的有效治疗措施。固醇调节元件结合蛋白(SREBP)是一类重要的核转录因子,主要通过激活胆固醇、脂肪酸和甘油三酯合成及摄取的相关基因来维持体内脂质代谢的平衡,是治疗代谢性疾病的靶点。本文对SREBP参与NAFLD发病机制的最新进展以及SREBP靶向治疗NAFLD的最新证据进行综述。值得注意的是,最近研究发现SREBP抑制与自噬受损共同引发肝损伤。因此,过度抑制脂肪生成对NAFLD的治疗可能起反作用。总体而言,SREBP是一个具有广阔前景的NAFLD治疗靶点,其对脂质代谢的分子机制受多种因素的调控,而这些因素正在被深入探索和分析,对NAFLD治疗具有重要的临床意义。
-
关键词:
- 非酒精性脂肪性肝病 /
- 胆固醇调节元件结合蛋白质类 /
- 脂肪生成 /
- 炎症 /
- 纤维化
Abstract: Nonalcoholic fatty liver disease (NAFLD) has become the most common liver disease in the world and is an important risk factor for the progression to hepatocellular carcinoma. However, the pathogenesis of NAFLD remains unclear, and there is still a lack of specific treatment measures. Sterol regulatory element-binding proteins (SREBP) are an important nuclear transcription factor, which mainly maintains the balance of lipid metabolism inside the body by activating the genes associated with the synthesis and uptake of cholesterol, fatty acids, and triglycerides, and therefore, SREBP are a target for the treatment of metabolic diseases. This article reviews the latest advances in SREBP in the pathogenesis of NAFLD and the latest evidence of SREBP-targeted therapy for NAFLD. It is worth noting that recent studies have shown that SREBP inhibition can cause liver injury together with autophagy damage. Therefore, excessive inhibition of lipogenesis may exert a counterproductive effect on the treatment of NAFLD. In conclusion, SREBP is a promising therapeutic target for NAFLD; the molecular mechanism of SREBP in lipid metabolism is regulated by many factors, and these factors are being deeply explored and analyzed, which has an important clinical significance for the treatment of NAFLD. -
非酒精性脂肪性肝病(NAFLD)是由肝脂肪堆积引起的一系列疾病,与代谢综合征密切相关[1]。NAFLD与肥胖和胰岛素抵抗相关,是目前最常见的影响不同年龄人群的慢性肝病[2]。临床中,NAFLD与多种肝脏异常相关,包括脂肪变性、非酒精性脂肪性肝炎(NASH)、纤维化和肝硬化[3]。运动和饮食是干预NAFLD的主要选择,但大多数患者依从性差,难以长期坚持。此外,目前尚无被批准治疗NAFLD的药物[4]。针对细胞、分子机制以及环境与遗传因素相互作用的研究已揭示出NAFLD发病机制中的众多关键靶点,为NAFLD的靶向治疗药物研发提供了多维方向。固醇调节元件结合蛋白(sterol regulatory-element binding protein,SREBP)是调节肝细胞脂质稳态的重要转录因子。研究[5]表明,SREBP参与NAFLD的发病机制,过度激活可促进NAFLD发生、发展。本文将阐述SREBP在NAFLD相关发病机制中的作用,归纳以SREBP为靶点的NAFLD治疗药物的研究进展。
1. SREBP家族是NAFLD脂质代谢的主要调控因子
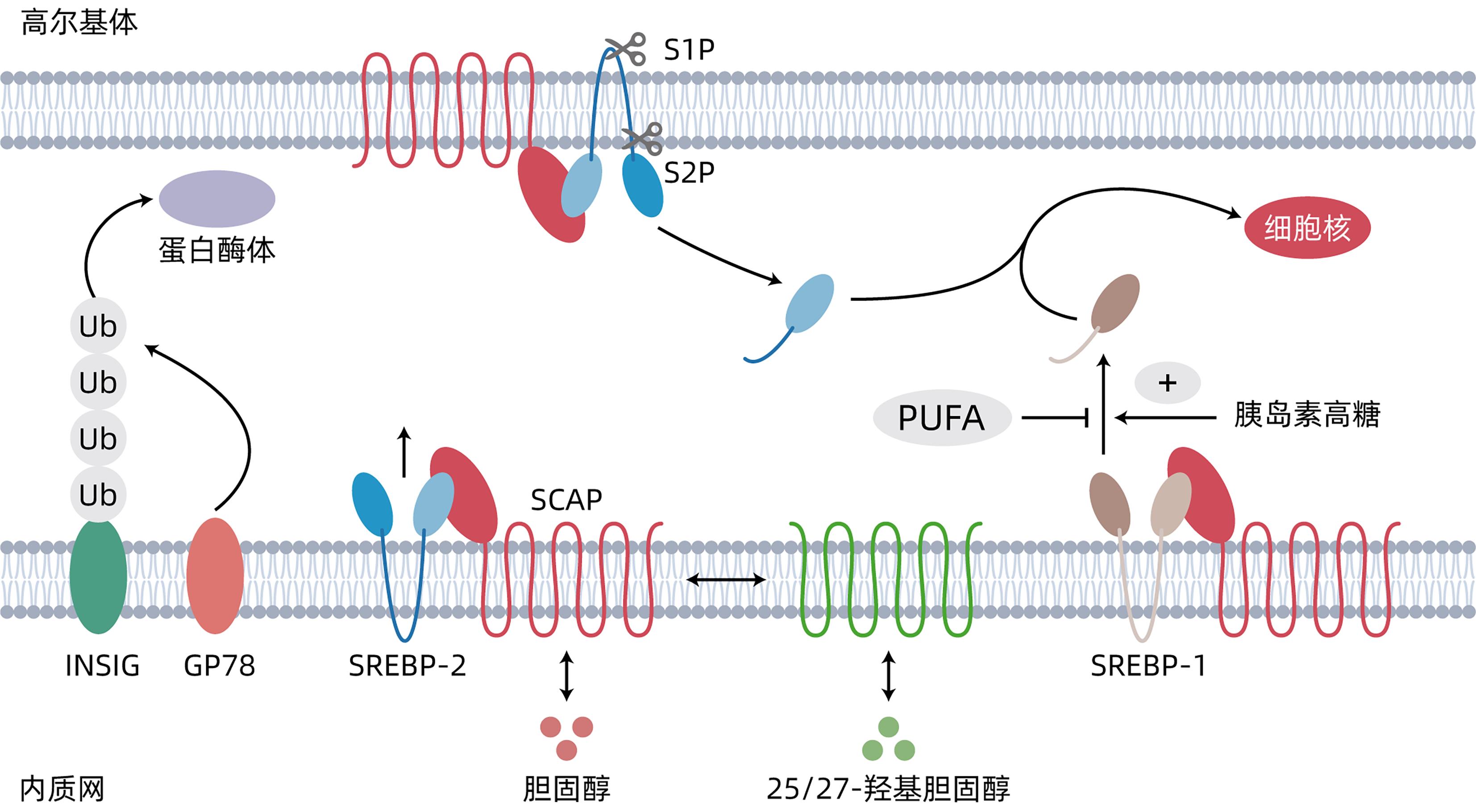
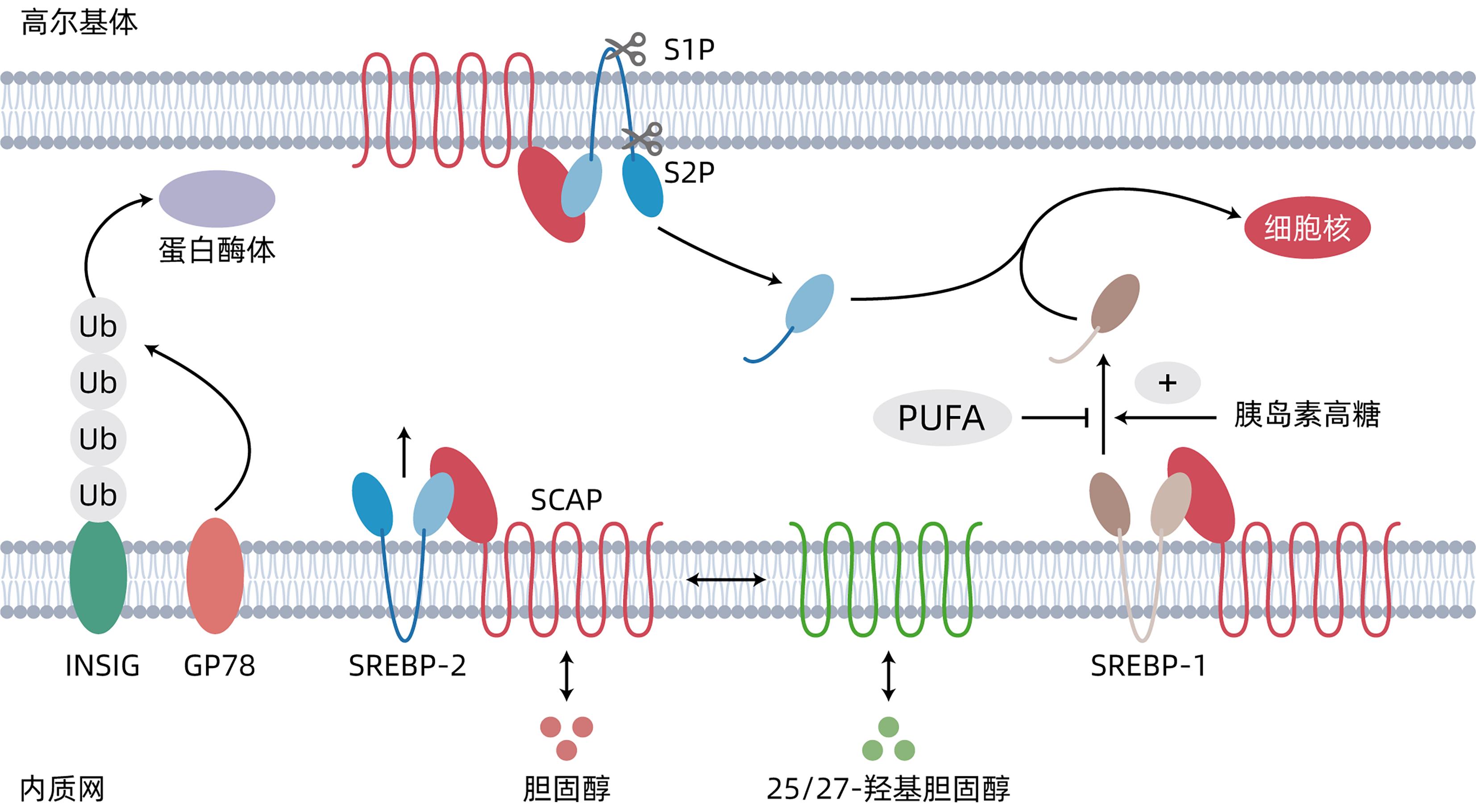
SREBP属于碱性-螺旋-环-螺旋亮氨酸拉链(bHLH-zip)家族。哺乳动物有3种SREBP亚型(SREBP-1a、SREBP-1c和SREBP-2),分别由SREBF1和SREBF2基因编码。在功能上,SREBP-1a和SREBP-1c参与脂肪酸代谢,而SREBP-2主要调节总胆固醇的生物合成[6]。但也有研究[7]发现,SREBP-1a不参与肝脂肪生成。SREBP起始定位于内质网膜上,并与SREBP裂解激活蛋白(SREBP-cleavage activating protein,SCAP)结合形成稳定的复合物,通过相同的加工途径被高尔基体中的位点1蛋白酶(site-1 protease,S1P)和S2P依次切割以形成成熟的SREBP,然后从高尔基体中释放出来并转运至细胞核中[8]。此外,胰岛素诱导基因1(insulin-induced gene 1,INSIG-1)和INSIG-2与SREBP-SCAP形成复合体,抑制SREBP向高尔基体的转运[9]。成熟的SREBP-1与固醇调节元件结合,促进多种脂肪生成基因的转录。SREBP异构体的基因表达和激活的调节机制也不相同,SREBP-1c受胰岛素和胰高血糖素的严格调节,而SREBP-2则受固醇水平的调节(图1)。尽管作用不同,但有证据[10]表明,SREBP-1c的缺失在一定程度上被SREBP-2补偿,表明2种异构体存在紧密的串扰和差异调节。
2. SREBP参与NAFLD的发病机制
2.1 脂肪生成
脂质代谢异常在早期NAFLD中发挥着关键作用。SREBP编码促进肝脂肪积累的酶,包括脂肪酸合成酶、乙酰辅酶A羧基酶(acetyl-CoA carboxylase,ACC)和3-羟基-3-甲基戊二酰辅酶A还原酶[11]。SREBP-1c是肝脂质从头合成的主要调节因子,可通过增加从头合成促进NAFLD[12]。
除了肝脏中甘油三酯的积累,肝内总胆固醇稳态的破坏也与NAFLD的发病机制有关。低密度脂蛋白受体(low-density lipoprotein receptor,LDLR)途径是一种负反馈系统,在血浆和细胞内总胆固醇稳态的调节中发挥重要作用[13]。LDLR通路失调可加剧NAFLD的进展。LDLR在转录水平上受SREBP-2和SCAP的严格调节,并在转录后水平上受到前蛋白转化酶枯草杆菌蛋白酶(recombinant proprotein convertase subtilisin/Kexin type 9,PCSK9)的严格调节[14]。SREBP-2异常上调引起LDLR大量摄取外源性胆固醇,同时显著提高内源性胆固醇的合成速率,导致总胆固醇代谢异常,这是NAFLD的重要病因之一。与LDLR相同,PCSK9主要在转录水平上受SREBP-2的调控[15]。然而,SREBP-2在总胆固醇过量的情况下仍可能被激活,从而使NASH患者炎症加剧,甚至导致细胞死亡[16]。
2.2 自噬、凋亡和炎症
自噬是一种细胞内降解系统,可将大分子和细胞器转运至溶酶体清除。此外,自噬还介导细胞脂质降解(称为脂噬)[17]。在NAFLD中,细胞内脂质异常升高可损害自噬功能,反之,自噬功能障碍通过引起内质网应激和增加胰岛素抵抗而加剧NAFLD。除了脂肪酸合成增加外,多种NAFLD致病因素也可损害肝脏自噬[18]。SREBP-1c可损害脂噬,促进肝脂肪变性[19]。Nguyen等[20]发现SREBF1通过激活miR-216a抑制脂质分解代谢,从而减少胱硫醚-γ-裂解酶-H2S信号传导和UNC-51样激酶1刺激的自噬。
当细胞处于极端的应激状态时,自噬或脂噬较大可能伴随细胞凋亡。SREBP参与细胞凋亡级联反应,例如凋亡抑制因子6作为巨噬细胞中SREBP-1a的靶点,SREBP-1a缺失可导致细胞凋亡加剧[21]。SREBP及其下游靶标在坏死性凋亡过程中诱导极长链脂肪酸的积累,加剧坏死性凋亡中质膜的通透性和细胞死亡[22]。
脂质作为一种炎症介质,在代谢、免疫和癌症中发挥着关键作用。SREBP可通过调节靶基因,影响巨噬细胞极化和吞噬作用,对炎症和免疫系统产生影响。SCAP-SREBP-2是巨噬细胞整合总胆固醇代谢和炎症的信号中枢,负责整合巨噬细胞中NOD样受体蛋白3炎症小体激活及总胆固醇合成信号传导[23]。
2.3 氧化应激
氧化应激是NAFLD肝损伤和疾病进展的重要调节因素。氧化应激可促进活性氧的产生,从而破坏氧化还原平衡,促进肝脂质积累。SREBP-1c是NAFLD中线粒体生物合成和氧化磷酸化功能的关键调节因子[24]。此外,SREBP-1c也是代谢重塑的关键调节因子,在热量限制诱导下的线粒体生物合成和减轻白色脂肪组织的氧化应激方面具有重要作用[25]。SREBP-2介导的固醇合成可通过减少脂质过氧化,保护细胞膜的完整性,从而对抗氧化应激[26]。
2.4 胰岛素抵抗
胰岛素抵抗是NAFLD的主要危险因素之一。在胰岛素抵抗状态下,来自脂肪细胞的脂肪酸流入增加,脂肪酸氧化减少,导致甘油三酯在肝脏中积累。胰岛素通过内质网启动的蛋白水解机制诱导SREBP-1c成熟。SREBP-1c在肝脏中的失控激活可直接抑制胰岛素信号通路,促进NAFLD的发展[27]。胰岛素从多个层面通路调控SREBP,包括SREBP mRNA的转录、SREBP的蛋白水解加工以及n-SREBP(成熟核型SREBP)的稳定性和丰度。胰岛素主要通过雷帕霉素靶蛋白C1(mammalian target of rapamycin C1,mTORC1)增强SREBP-1c mRNA的转录。mTORC1-p70核糖体蛋白S6激酶(p70 ribosomal protein S6 kinase 1,S6K1)对于胰岛素抵抗条件下SREBP激活以及持续脂肪生成和肝脂肪变性至关重要。SREBP-1和SREBP-2是促进S6K1介导的细胞增殖的重要节点[28]。在NAFLD患者中,胰岛素激活胰岛素受体底物(insulin receptor substrate,IRS)蛋白,通过肝脏表达SREBP-1c,刺激脂肪生成,导致肝细胞内脂肪酸合成增加[29]。上述过程可导致恶性循环,即胰岛素抵抗增加新的脂质合成,脂质积累又加重胰岛素抵抗。相反,SREBP-1c通过抑制肝脏IRS2信号诱导胰岛素抵抗[30]。
2.5 PUFA和氨基酸
PUFA在体内抑制SREBP-1c,而非SREBP-2。PUFA通过多种机制抑制SREBP-1c,例如抑制SREBP-1c转录、核转位以及蛋白水解裂解和蛋白酶体降解,并增强其mRNA降解[31]。因此,PUFA有助于减少肝脂肪堆积,从而预防NAFLD。n-3 PUFA是一种长链脂肪酸,能够抑制SREBP-1c的活性,增加肝脂肪酸氧化,降低甘油三酯合成[32]。
氨基酸水平也可激活和调节SREBP的表达。氨基酸可激活溶酶体中的mTORC1并调节蛋白质合成和自噬。丝氨酸/苏氨酸蛋白激酶2是氨基酸缺乏感受器,能够抑制蛋白质翻译,在营养缺乏时降低SREBP-1和脂肪生成酶的水平。在禁食或氨基酸缺乏的情况下,SREBP-1c通过丝氨酸/苏氨酸蛋白激酶2或mTOR途径受到抑制。有研究[33]利用等压串联质量标签和质谱,在大鼠SREBP-1c中鉴定出4个丝氨酸残基(S418、S419、S422和S424),这些丝氨酸残基在胰岛素作用下被特异性磷酸化,是pSREBP-1c中p70S6激酶磷酸化的关键靶点。同时,研究还发现pSREBP-1c在胰岛素作用下的磷酸化很可能是由p70S6激酶介导的,这一过程反之又会提高nSREBP-1c对参与脂质合成基因的激活潜力。
2.6 内质网应激
内质网应激可直接或间接上调SREBP,促进肝脂肪变性的发生,从而参与NAFLD的发展[34]。由于ATF6(转录激活因子6,内质网应激近端传感器之一)和SREBP-1的激活均由相同的蛋白酶在相同的位置诱导,故内质网应激与SREBP激活存在紧密联系[35]。在内质网应激期间,PERK-IRE1-eIF2α-ATF6信号通路的慢性激活导致SREBP-1c的过度激活和肝脂肪变性[36-37]。
2.7 昼夜节律
昼夜节律与细胞和器官的代谢密切相关。昼夜节律紊乱可加速NAFLD进展,同时NAFLD也可破坏昼夜节律。SREBP与时钟基因编码的蛋白质在昼夜节律调节中相互作用[38]。REV-ERBα是昼夜节律周期的重要反馈调节因子,可调节SREBP活性,在总胆固醇和胆汁酸合成的昼夜节律调节中发挥关键作用[39]。最近研究[40]表明,肝细胞SREBP信号还介导肝脏内的时钟通讯。为了维持稳定的胆固醇水平,胆固醇的昼夜利用必须通过胆固醇合成和摄取实现平衡,SREBP的固醇依赖性加工和激活或可满足这一目标。
进一步而言,非核心时钟转录因子也可作为代谢的昼夜调节因子。最近研究[41]表明,肠道时钟可重编程肝脏节律性代谢。肠道特异性脑和肌肉芳香烃受体核转运样蛋白1基因敲除可通过肠道传递的PUFA对肝脏SREBP-1c产生节律性破坏,导致肝脏中脂质和葡萄糖的昼夜代谢稳态被破坏。
3. 靶向SREBP治疗NAFLD
SREBP-1c在NAFLD的发病机制中发挥重要作用。抑制SREBP途径可减少脂质生成,从而降低NAFLD风险。SREBP的小分子抑制剂是开发有效治疗代谢性疾病的重要工具。然而,目前尚无针对SREBP的药物上市,因此迫切需要开发高特异性的SREBP小分子抑制剂。
3.1 合成小分子药物
法图他汀是第1个被发现能够抑制SREBP激活过程的非固醇类小分子,能够直接结合SCAP并阻止其从内质网转运至高尔基体,从而抑制SREBP的激活[42]。
哺乳动物不育系20-样激酶1(mammalian sterile 20-like kinase1,MST1)是一种丝氨酸/苏氨酸蛋白激酶,通过减少从头合成和加速脂肪酸氧化,显著减少NAFLD小鼠肝脏中的脂质积聚[43]。2022年,有研究[44]报道一种基于肝素多糖的PGEA聚阳离子载体(Hep@PGEA),基于Hep@PGEA的MST1(HCP/MST1)基因传递系统,通过调节腺苷酸活化蛋白激酶(adenosine activated protein kinase,AMPK)/SREBP-1c信号通路,抑制SREBP-1c的转录和脂肪酸合成酶(fatty acid synthase,FAS)的表达,可见显著提高肝脏胰岛素敏感性,减少肝损伤和肝脂质积累,对NAFLD的治疗颇有益处且无明显毒性。
25-羟基维生素D也是一种SREBP抑制剂,可通过诱导蛋白水解加工和泛素介导的SCAP降解,损害SREBP的激活[45]。Kawagoe等[46]利用一个完全非天然的实体取代25-羟基维生素D3的A环,开发出可减轻小鼠肝脂质积累而不产生钙化作用的KK-052。
诱饵寡脱氧核苷酸技术可在DNA水平上阻断mRNA转录[47]。SREBP诱饵寡脱氧核苷酸是一种新型SREBP抑制剂,通过抑制SREPB信号通路调节脂质代谢并抑制脂肪生成,从而减少肝脂质积累[48]。
近期,Jiang等[49]发现一个改善NASH的新型化合物:25-羟基羊毛固醇,是一个强效且特异性的SREBP通路抑制剂,通过靶向INSIG蛋白以阻止SREBP激活,且相较于奥贝胆酸对NASH的治疗更为有效。
3.2 天然小分子药物
除了白桦脂醇、黄腐酚和大黄素等经典的SREBP天然抑制剂,近期也发现许多新型SREBP抑制剂。
最近研究[50]表明,含有大黄游离蒽醌的二氯甲烷提取物可通过显著下调SREBP-2 mRNA表达以及增加过氧化物酶体增殖物激活受体α的表达,下调硬脂酰辅酶A去饱和酶-1的表达,并减少3-羟基-3-甲基戊二酰辅酶A还原酶的表达,从而降低肝脏和血清总胆固醇水平。Su等[51]发现芦荟大黄素选择性抑制SREBP-2和肝细胞核因子1α介导的PCSK9信号传导,从而上调LDLR并促进LDL摄取。
萝卜硫素(sulforaphane,SFaN)是一种新发现的SREBP天然抑制剂,其通过促进SREBP前体形式降解,抑制SREBP活性。此外,异硫氰酸盐SFaN和SFeN可抑制FAS基因启动子活性和SREBP靶基因表达[52]。
异槲皮苷是一种在槲皮苷第3位具有糖苷键的多酚,可提取于芒果和大黄等多种植物[53]。最近研究[54]表明,异槲皮苷通过靶向AMPK/ACC和SREBP-1/FAS途径调节脂质代谢,改善NAFLD。异槲皮苷激活AMPK磷酸化ACC,抑制SREBP-1/FAS信号,从而抑制脂质生成。
3.3 微小RNA(microRNA,miRNA)对脂质代谢的调控
除了脂质代谢的经典转录调节,miRNA也是转录后水平脂质代谢的有效调节因子。miRNA的异常表达与NAFLD有关,部分miRNA(例如由SREBP-2控制的miR-33a)可抑制高密度脂蛋白形成,另有部分miRNA(例如由SREBP-1控制的miR-33b)抑制脂肪酸代谢。直接、特异性靶向SREBP的miRNA可能是新的NAFLD治疗策略的未来方向。
脂质代谢领域中被研究最多的miRNA是miR-33a。miR-33a位于SREBF2的内含子中,可通过下调ABC转运蛋白的表达,调节高密度脂蛋白生物合成和总胆固醇流出[55]。miR-33a与SREBP-2协同作用,在维持细胞内总胆固醇稳态中发挥重要作用[56]。此外,miR-33反义靶向可能会逆转IRS2和IRS1在肝脏中的表达变化,从而降低SREBP-1c的水平。
Li等[57]研究证实,miR-23a/b-3p可通过结合SREBP-1c和FAS mRNA的5'-UTR,增强mRNA的稳定性,从而促进甘油三酯在肝细胞中的积累。近期研究[58]表明,miR-615-5p能够在mRNA和蛋白质水平上显著降低mTOR和SREBP-1c,从而减少肝细胞脂滴形成,并降低甘油三酯积累。而长链非编码RNA-H19仅在蛋白质水平上降低这2个靶点。综上所述,miRNA疗法在未来NAFLD的治疗中具有广阔前景。
3.4 对SREBP的其他调节
近几十年来,SREBP通路及其调控机制逐渐被认识。因此,推测SREBP抑制剂可能在以下几个方面发挥作用:(1)抑制SREBP基因的表达;(2)刺激SCAP和INSIG之间的相互作用,将前体SREBP锚定在内质网上;(3)阻断SREBP-SCAP复合物从内质网向高尔基体的迁移;(4)抑制2种位点特异性蛋白酶S1P和S2P;(5)阻止成熟SREBP进入细胞核;(6)加速N端SREBP降解;(7)miRNA。针对以上靶点机制进行SREBP抑制剂的开发,对于NAFLD的治疗可发挥事半功倍的作用。
调节SREBP以恢复异常的自噬功能可能是防治NAFLD的一个潜在的新方向。Cheng等[59]研究发现,SREBP-2可激活不同肝细胞中的自噬相关基因,且SREBP-2的过度表达部分缓解了脂质过量的肝细胞中受抑制的自噬活性。在体内外NAFLD小鼠模型研究[60]中证实,抑制SREBP-2的核转位可改善受损的自噬通量,并减轻内质网应激。
拟素化修饰是SREBP-1c的一种新的翻译后修饰。拟素化修饰通过阻断SREBP-1c的泛素化而增强SREBP-1c介导的肝脂肪生成,拟素化抑制剂MLN4924通过阻断SREBP-1c拟素化修饰,降低脂质基因表达和肝脏甘油三酯,对NAFLD具有预防和治疗作用[61]。因此,阻断SREBP-1c拟素化修饰也可能是一种治疗NAFLD的方法。
肝纤维化是NAFLD晚期最重要的病理改变,其特征是细胞外基质的积累。SREBP-1c可促进肝脂肪变性,然而其可能也是肝纤维化的负调控因子。Su等[62]研究证实SREBP-1c对肝纤维化具有抑制作用,其机制与SREBP-1对转化生长因子β1水平、转化生长因子β1受体和血小板源性生长因子β受体及其下游信号传导的影响以及基因表观遗传调控分子有关。因此,针对肝星状细胞中的SREBP可能是NASH的独特治疗策略。
4. 小结与展望
过度激活或抑制SREBP均可能导致NAFLD疾病进展,因此维持适当的SREBP活性可能会更有效地治疗NAFLD。例如,Kawamura等[63]研究发现,强烈抑制SCAP/SREBP介导的脂肪生成扰乱磷脂代谢,可加剧小鼠NASH的肝损伤、纤维化和癌变,且完全抑制SREBP活性会破坏细胞膜脂质稳态并诱导内质网应激,不仅无法改善疾病,反而加速了NASH和肝细胞癌的进展,这说明通过SCAP/SREBP途径直接或间接靶向脂肪生成的治疗策略还不够成熟和完善。SREBP需要适当的调节以维持能量状态、生长信号和器官稳态。因此,SREBP也可以被称作“固醇调节的生物学程序”。
NAFLD是一种代谢性疾病,对多个系统产生影响。因此,NAFLD的治疗策略不应局限于以肝脏为中心,而是应从多学科中获得综合治疗策略。SREBP作为脂质合成的关键调节因子,将其作为靶点的新型药物的研发也引起人们的重视。NAFLD的发生不仅涉及肝脏的分子变化,还涉及器官间的代谢信号。因此,还需要在后续研究中深入研究SREBP对NAFLD的调控作用,这将对SREBP抑制剂的研发大有帮助,对未来更加安全、高效、低毒的药物研发也能够起到一定的指导作用,并为进一步改善NAFLD的治疗奠定坚实的基础。
-
[1] MUNDI MS, VELAPATI S, PATEL J, et al. Evolution of NAFLD and its management[J]. Nutr Clin Pract, 2020, 35( 1): 72- 84. DOI: 10.1002/ncp.10449. [2] KAUFMANN B, RECA A, WANG B, et al. Mechanisms of nonalcoholic fatty liver disease and implications for surgery[J]. Langenbecks Arch Surg, 2021, 406( 1): 1- 17. DOI: 10.1007/s00423-020-01965-1. [3] POUWELS S, SAKRAN N, GRAHAM Y, et al. Non-alcoholic fatty liver disease(NAFLD): A review of pathophysiology, clinical management and effects of weight loss[J]. BMC Endocr Disord, 2022, 22( 1): 63. DOI: 10.1186/s12902-022-00980-1. [4] FRIEDMAN SL, NEUSCHWANDER-TETRI BA, RINELLA M, et al. Mechanisms of NAFLD development and therapeutic strategies[J]. Nat Med, 2018, 24( 7): 908- 922. DOI: 10.1038/s41591-018-0104-9. [5] XU XH, POULSEN KL, WU LJ, et al. Targeted therapeutics and novel signaling pathways in non-alcohol-associated fatty liver/steatohepatitis(NAFL/NASH)[J]. Signal Transduct Target Ther, 2022, 7( 1): 287. DOI: 10.1038/s41392-022-01119-3. [6] GOLDSTEIN JL, DEBOSE-BOYD RA, BROWN MS. Protein sensors for membrane sterols[J]. Cell, 2006, 124( 1): 35- 46. DOI: 10.1016/j.cell.2005.12.022. [7] ARAKI M, NAKAGAWA Y, SAITO H, et al. Hepatocyte- or macrophage-specific SREBP-1a deficiency in mice exacerbates methionine- and choline-deficient diet-induced nonalcoholic fatty liver disease[J]. Am J Physiol Gastrointest Liver Physiol, 2022, 323( 6): G627- G639. DOI: 10.1152/ajpgi.00090.2022. [8] SATO R. Sterol metabolism and SREBP activation[J]. Arch Biochem Biophys, 2010, 501( 2): 177- 181. DOI: 10.1016/j.abb.2010.06.004. [9] NAKAGAWA Y, ARAKI M, HAN SI, et al. CREBH systemically regulates lipid metabolism by modulating and integrating cellular functions[J]. Nutrients, 2021, 13( 9): 3204. DOI: 10.3390/nu13093204. [10] JEON YG, KIM YY, LEE G, et al. Physiological and pathological roles of lipogenesis[J]. Nat Metab, 2023, 5( 5): 735- 759. DOI: 10.1038/s42255-023-00786-y. [11] MARGERIE D, LEFEBVRE P, RAVERDY V, et al. Hepatic transcriptomic signatures of statin treatment are associated with impaired glucose homeostasis in severely obese patients[J]. BMC Med Genomics, 2019, 12( 1): 80. DOI: 10.1186/s12920-019-0536-1. [12] IPSEN DH, LYKKESFELDT J, TVEDEN-NYBORG P. Molecular mechanisms of hepatic lipid accumulation in non-alcoholic fatty liver disease[J]. Cell Mol Life Sci, 2018, 75( 18): 3313- 3327. DOI: 10.1007/s00018-018-2860-6. [13] GOLDSTEIN JL, BASU SK, BROWN MS. Receptor-mediated endocytosis of low-density lipoprotein in cultured cells[J]. Methods Enzymol, 1983, 98: 241- 260. DOI: 10.1016/0076-6879(83)98152-1. [14] NOHTURFFT A, DEBOSE-BOYD RA, SCHEEK S, et al. Sterols regulate cycling of SREBP cleavage-activating protein(SCAP) between endoplasmic reticulum and Golgi[J]. Proc Natl Acad Sci U S A, 1999, 96( 20): 11235- 11240. DOI: 10.1073/pnas.96.20.11235. [15] HORTON JD, COHEN JC, HOBBS HH. Molecular biology of PCSK9: Its role in LDL metabolism[J]. Trends Biochem Sci, 2007, 32( 2): 71- 77. DOI: 10.1016/j.tibs.2006.12.008. [16] MUSSO G, GAMBINO R, CASSADER M. Cholesterol metabolism and the pathogenesis of non-alcoholic steatohepatitis[J]. Prog Lipid Res, 2013, 52( 1): 175- 191. DOI: 10.1016/j.plipres.2012.11.002. [17] CAROTTI S, AQUILANO K, ZALFA F, et al. Lipophagy impairment is associated with disease progression in NAFLD[J]. Front Physiol, 2020, 11: 850. DOI: 10.3389/fphys.2020.00850. [18] ALLAIRE M, RAUTOU PE, CODOGNO P, et al. Autophagy in liver diseases: Time for translation?[J]. J Hepatol, 2019, 70( 5): 985- 998. DOI: 10.1016/j.jhep.2019.01.026. [19] NGUYEN TTP, KIM DY, LEE YG, et al. SREBP-1c impairs ULK1 sulfhydration-mediated autophagic flux to promote hepatic steatosis in high-fat-diet-fed mice[J]. Mol Cell, 2021, 81( 18): 3820- 3832. DOI: 10.1016/j.molcel.2021.06.003. [20] NGUYEN TTP, KIM DY, IM SS, et al. Impairment of ULK1 sulfhydration-mediated lipophagy by SREBF1/SREBP-1c in hepatic steatosis[J]. Autophagy, 2021, 17( 12): 4489- 4490. DOI: 10.1080/15548627.2021.1968608. [21] IM SS, OSBORNE TF. Protection from bacterial-toxin-induced apoptosis in macrophages requires the lipogenic transcription factor sterol regulatory element binding protein 1a[J]. Mol Cell Biol, 2012, 32( 12): 2196- 2202. DOI: 10.1128/MCB.06294-11. [22] LU D, PARISI LR, GOKCUMEN O, et al. SREBP activation contributes to fatty acid accumulations in necroptosis[J]. RSC Chem Biol, 2023, 4( 4): 310- 322. DOI: 10.1039/d2cb00172a. [23] GUO CS, CHI ZX, JIANG DL, et al. Cholesterol homeostatic regulator SCAP-SREBP2 integrates NLRP3 inflammasome activation and cholesterol biosynthetic signaling in macrophages[J]. Immunity, 2018, 49( 5): 842- 856. DOI: 10.1016/j.immuni.2018.08.021. [24] PETTINELLI P, DEL POZO T, ARAYA J, et al. Enhancement in liver SREBP-1c/PPAR-alpha ratio and steatosis in obese patients: Correlations with insulin resistance and n-3 long-chain polyunsaturated fatty acid depletion[J]. Biochim Biophys Acta, 2009, 1792( 11): 1080- 1086. DOI: 10.1016/j.bbadis.2009.08.015. [25] FUJII N, NARITA T, OKITA N, et al. Sterol regulatory element-binding protein-1c orchestrates metabolic remodeling of white adipose tissue by caloric restriction[J]. Aging Cell, 2017, 16( 3): 508- 517. DOI: 10.1111/acel.12576. [26] SOUNDARARAJAN R, WISHART AD, VASANTHA RUPASINGHE HP, et al. Quercetin 3-glucoside protects neuroblastoma(SH-SY5Y) cells in vitro against oxidative damage by inducing sterol regulatory element-binding protein-2-mediated cholesterol biosynthesis[J]. J Biol Chem, 2008, 283( 4): 2231- 2245. DOI: 10.1074/jbc.M703583200. [27] SHIMANO H, SATO R. SREBP-regulated lipid metabolism: Convergent physiology-divergent pathophysiology[J]. Nat Rev Endocrinol, 2017, 13( 12): 710- 730. DOI: 10.1038/nrendo.2017.91. [28] DÜVEL K, YECIES JL, MENON S, et al. Activation of a metabolic gene regulatory network downstream of mTOR complex 1[J]. Mol Cell, 2010, 39( 2): 171- 183. DOI: 10.1016/j.molcel.2010.06.022. [29] KOHJIMA M, HIGUCHI N, KATO M, et al. SREBP-1c, regulated by the insulin and AMPK signaling pathways, plays a role in nonalcoholic fatty liver disease[J]. Int J Mol Med, 2008, 21( 4): 507- 511. [30] PUIGSERVER P, RHEE J, DONOVAN J, et al. Insulin-regulated hepatic gluconeogenesis through FOXO1-PGC-1alpha interaction[J]. Nature, 2003, 423( 6939): 550- 555. DOI: 10.1038/nature01667. [31] JUMP DB. Dietary polyunsaturated fatty acids and regulation of gene transcription[J]. Curr Opin Lipidol, 2002, 13( 2): 155- 164. DOI: 10.1097/00041433-200204000-00007. [32] GNONI A, GIUDETTI AM. Dietary long-chain unsaturated fatty acids acutely and differently reduce the activities of lipogenic enzymes and of citrate carrier in rat liver[J]. J Physiol Biochem, 2016, 72( 3): 485- 494. DOI: 10.1007/s13105-016-0495-3. [33] DONG QM, MAJUMDAR G, O’MEALLY RN, et al. Insulin-induced de novo lipid synthesis occurs mainly via mTOR-dependent regulation of proteostasis of SREBP-1c[J]. Mol Cell Biochem, 2020, 463( 1-2): 13- 31. DOI: 10.1007/s11010-019-03625-5. [34] FLISTER KFT, PINTO BAS, FRANÇA LM, et al. Long-term exposure to high-sucrose diet down-regulates hepatic endoplasmic reticulum-stress adaptive pathways and potentiates de novo lipogenesis in weaned male mice[J]. J Nutr Biochem, 2018, 62: 155- 166. DOI: 10.1016/j.jnutbio.2018.09.007. [35] YE J, RAWSON RB, KOMURO R, et al. ER stress induces cleavage of membrane-bound ATF6 by the same proteases that process SREBPs[J]. Mol Cell, 2000, 6( 6): 1355- 1364. DOI: 10.1016/s1097-2765(00)00133-7. [36] LEE JS, ZHENG Z, MENDEZ R, et al. Pharmacologic ER stress induces non-alcoholic steatohepatitis in an animal model[J]. Toxicol Lett, 2012, 211( 1): 29- 38. DOI: 10.1016/j.toxlet.2012.02.017. [37] RÖHRL C, EIGNER K, WINTER K, et al. Endoplasmic reticulum stress impairs cholesterol efflux and synthesis in hepatic cells[J]. J Lipid Res, 2014, 55( 1): 94- 103. DOI: 10.1194/jlr.M043299. [38] GILARDI F, MIGLIAVACCA E, NALDI A, et al. Genome-wide analysis of SREBP1 activity around the clock reveals its combined dependency on nutrient and circadian signals[J]. PLoS Genet, 2014, 10( 3): e1004155. DOI: 10.1371/journal.pgen.1004155. [39] MARTELOT GL, CLAUDEL T, GATFIELD D, et al. REV-ERBalpha participates in circadian SREBP signaling and bile acid homeostasis[J]. PLoS Biol, 2009, 7( 9): e1000181. DOI: 10.1371/journal.pbio.1000181. [40] GUAN DY, BAE H, ZHOU DS, et al. Hepatocyte SREBP signaling mediates clock communication within the liver[J]. J Clin Invest, 2023, 133( 8): e163018. DOI: 10.1172/JCI163018. [41] CHEN M, LIN YK, DANG YK, et al. Reprogramming of rhythmic liver metabolism by intestinal clock[J]. J Hepatol, 2023, 79( 3): 741- 757. DOI: 10.1016/j.jhep.2023.04.040. [42] SHAO W, MACHAMER CE, ESPENSHADE PJ. Fatostatin blocks ER exit of SCAP but inhibits cell growth in a SCAP-independent manner[J]. J Lipid Res, 2016, 57( 8): 1564- 1573. DOI: 10.1194/jlr.M069583. [43] LI YH, LUAN YS, LI JN, et al. Exosomal miR-199a-5p promotes hepatic lipid accumulation by modulating MST1 expression and fatty acid metabolism[J]. Hepatol Int, 2020, 14( 6): 1057- 1074. DOI: 10.1007/s12072-020-10096-0. [44] LI YH, NIE JJ, YANG YH, et al. Redox-unlockable nanoparticle-based MST1 delivery system to attenuate hepatic steatosis via the AMPK/SREBP-1c signaling axis[J]. ACS Appl Mater Interfaces, 2022, 14( 30): 34328- 34341. DOI: 10.1021/acsami.2c05889. [45] ASANO L, WATANABE M, RYODEN Y, et al. Vitamin D metabolite, 25-hydroxyvitamin D, regulates lipid metabolism by inducing degradation of SREBP/SCAP[J]. Cell Chem Biol, 2017, 24( 2): 207- 217. DOI: 10.1016/j.chembiol.2016.12.017. [46] KAWAGOE F, MENDOZA A, HAYATA Y, et al. Discovery of a vitamin D receptor-silent vitamin D derivative that impairs sterol regulatory element-binding protein in vivo[J]. J Med Chem, 2021, 64( 9): 5689- 5709. DOI: 10.1021/acs.jmedchem.0c02179. [47] ISHIBASHI H, NAKAGAWA K, ONIMARU M, et al. Sp1 decoy transfected to carcinoma cells suppresses the expression of vascular endothelial growth factor, transforming growth factor beta1, and tissue factor and also cell growth and invasion activities[J]. Cancer Res, 2000, 60( 22): 6531- 6536. [48] AN HJ, KIM JY, GWON MG, et al. Beneficial effects of SREBP decoy oligodeoxynucleotide in an animal model of hyperlipidemia[J]. Int J Mol Sci, 2020, 21( 2): 552. DOI: 10.3390/ijms21020552. [49] JIANG SY, YANG XL, YANG ZM, et al. Discovery of an insulin-induced gene binding compound that ameliorates nonalcoholic steatohepatitis by inhibiting sterol regulatory element-binding protein-mediated lipogenesis[J]. Hepatology, 2022, 76( 5): 1466- 1481. DOI: 10.1002/hep.32381. [50] WANG YD, ZHANG JN, XU Z, et al. Identification and action mechanism of lipid regulating components from Rhei Radix et rhizoma[J]. J Ethnopharmacol, 2022, 292: 115179. DOI: 10.1016/j.jep.2022.115179. [51] SU ZL, HANG PZ, HU J, et al. Aloe-emodin exerts cholesterol-lowering effects by inhibiting proprotein convertase subtilisin/kexin type 9 in hyperlipidemic rats[J]. Acta Pharmacol Sin, 2020, 41( 8): 1085- 1092. DOI: 10.1038/s41401-020-0392-8. [52] MIYATA S, KODAKA M, KIKUCHI A, et al. Sulforaphane suppresses the activity of sterol regulatory element-binding proteins(SREBPs) by promoting SREBP precursor degradation[J]. Sci Rep, 2022, 12( 1): 8715. DOI: 10.1038/s41598-022-12347-6. [53] CHO WK, LEE MM, MA JY. Antiviral effect of isoquercitrin against influenza A viral infection via modulating hemagglutinin and neuraminidase[J]. Int J Mol Sci, 2022, 23( 21): 13112. DOI: 10.3390/ijms232113112. [54] KIM SH, YUN C, KWON D, et al. Effect of isoquercitrin on free fatty acid-induced lipid accumulation in HepG2 cells[J]. Molecules, 2023, 28( 3): 1476. DOI: 10.3390/molecules28031476. [55] NAJAFI-SHOUSHTARI SH, KRISTO F, LI YX, et al. MicroRNA-33 and the SREBP host genes cooperate to control cholesterol homeostasis[J]. Science, 2010, 328( 5985): 1566- 1569. DOI: 10.1126/science.1189123. [56] HORIE T, NISHINO T, BABA O, et al. MicroRNA-33 regulates sterol regulatory element-binding protein 1 expression in mice[J]. Nat Commun, 2013, 4: 2883. DOI: 10.1038/ncomms3883. [57] LI LF, ZHANG XY, REN HJ, et al. MiR-23a/b-3p promotes hepatic lipid accumulation by regulating Srebp-1c and Fas[J]. J Mol Endocrinol, 2021, 68( 1): 35- 49. DOI: 10.1530/JME-20-0324. [58] SOBKY SA EL, ABOUD NK, ASSALY NM EL, et al. Regulation of lipid droplet(LD) formation in hepatocytes via regulation of SREBP1c by non-coding RNAs[J]. Front Med, 2022, 9: 903856. DOI: 10.3389/fmed.2022.903856. [59] CHENG CW, DENG XL, XU KS. Increased expression of sterol regulatory element binding protein-2 alleviates autophagic dysfunction in NAFLD[J]. Int J Mol Med, 2018, 41( 4): 1877- 1886. DOI: 10.3892/ijmm.2018.3389. [60] DENG XL, PAN XL, CHENG CW, et al. Regulation of SREBP-2 intracellular trafficking improves impaired autophagic flux and alleviates endoplasmic reticulum stress in NAFLD[J]. Biochim Biophys Acta Mol Cell Biol Lipids, 2017, 1862( 3): 337- 350. DOI: 10.1016/j.bbalip.2016.12.007. [61] JU UI, JEONG DW, SEO J, et al. Neddylation of sterol regulatory element-binding protein 1c is a potential therapeutic target for nonalcoholic fatty liver treatment[J]. Cell Death Dis, 2020, 11( 4): 283. DOI: 10.1038/s41419-020-2472-6. [62] SU SY, TIAN HM, JIA X, et al. Mechanistic insights into the effects of SREBP1c on hepatic stellate cell and liver fibrosis[J]. J Cell Mol Med, 2020, 24( 17): 10063- 10074. DOI: 10.1111/jcmm.15614. [63] KAWAMURA S, MATSUSHITA Y, KUROSAKI S, et al. Inhibiting SCAP/SREBP exacerbates liver injury and carcinogenesis in murine nonalcoholic steatohepatitis[J]. J Clin Invest, 2022, 132( 11): e151895. DOI: 10.1172/JCI151895. -



 PDF下载 ( 914 KB)
PDF下载 ( 914 KB)


 下载:
下载:
 下载:
下载: