组蛋白去乙酰化酶1对非酒精性脂肪性肝病细胞模型胰岛素抵抗的影响
DOI: 10.3969/j.issn.1001-5256.2022.09.013
Influence of histone deacetylase 1 on insulin resistance in a cell model of nonalcoholic fatty liver disease
-
摘要:
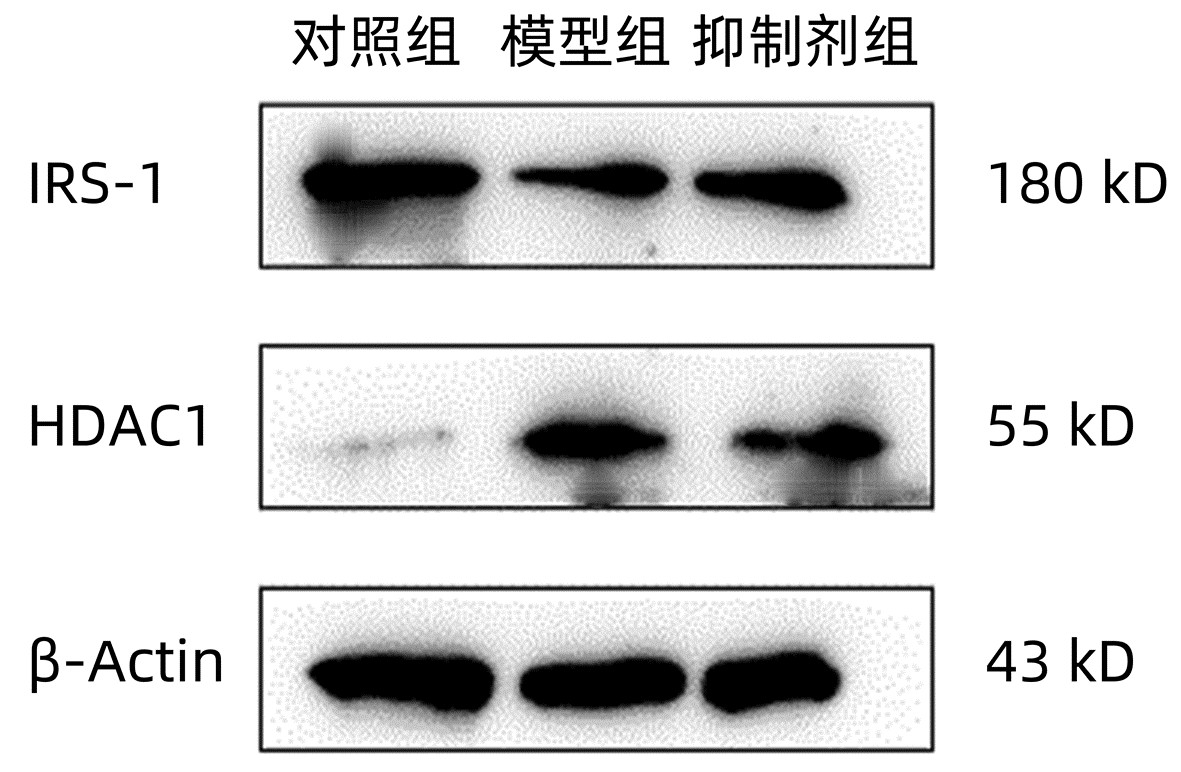
目的 建立高脂诱导的HepG2非酒精性脂肪性肝病(NAFLD)细胞模型,分析组蛋白去乙酰化酶1(HDAC1)表达对NAFLD细胞胰岛素抵抗(IR)的促进作用。 方法 将HepG2细胞分为对照组、模型组(OA)和抑制剂组[OA+Pyroxamide(HDAC1抑制剂)]。采用CCK-8法绘制HepG2细胞标准生长曲线,并筛选OA和Pyroxamide的最佳药物作用浓度和作用时间;油红O染色比较细胞内脂滴蓄积程度;全自动生化仪检测细胞内ALT、AST、TG、TC含量;RT-qPCR法和Western Blot法分别检测细胞内HDAC1、胰岛素受体底物-1(IRS-1)mRNA和蛋白表达量。计量资料多组间比较采用单因素方差分析,进一步两两比较采用LSD-t检验。 结果 0.25 mmol/L OA处理24 h为细胞最佳造模浓度和持续时间,20 μmol/L处理24 h为Pyroxamide最佳给药浓度和持续时间;模型组相比于对照组ALT、AST、TG、TC含量均明显升高,而抑制剂组相比于模型组ALT、AST、TG、TC含量均显著降低(P值均<0.05)。各组细胞内HDAC1 mRNA和蛋白表达量比较,模型组高于对照组,抑制剂组低于模型组,差异均有统计学意义(P值均<0.05);各组细胞内IRS-1 mRNA和蛋白表达量相比,模型组低于对照组,抑制剂组高于模型组,差异均有统计学意义(P值均<0.05)。 结论 HDAC1通过抑制IRS-1分子的表达,促进IR,参与了NAFLD的发生与发展,HDAC1抑制剂Pyroxamide通过减轻IR,对肝脏起保护作用。 Abstract:Objective To investigate the promoting effect of histone deacetylase 1 (HDAC1) expression on insulin resistance (IR) in nonalcoholic fatty liver disease (NAFLD) cells by establishing an HepG2 cell model of high fat-induced NAFLD. Methods HepG2 cells were divided into control group, model group (OA), and inhibitor group (OA+pyroxamide [an HDAC1 inhibitor]). CCK-8 assay was used to plot the standard growth curve of HepG2 cells and screen out the optimal drug concentration and action time of OA and pyroxamide; oil red O staining was used to compare the accumulation of lipid droplets in cells; an automatic biochemical analyzer was used to analyze the content of alanine aminotransferase (ALT), aspartate aminotransferase (AST), triglyceride (TG), and total cholesterol (TC) in cells; quantitative real-time PCR and Western blot were used to measure the mRNA and protein expression levels of HDAC1 and insulin receptor substrate-1 (IRS-1) in cells. A one-way analysis of variance was used for comparison of continuous data between multiple groups, and the least significant difference t-test was used for further comparison between two groups. Results OA treatment at a concentration of 0.25 mmol/L for 24 hours was the optimal concentration and duration of cell modeling, and treatment at a concentration of 20 μmol/L for 24 hours was the optimal administration concentration and duration of pyroxamide. Compared with the control group, the model group had significant increases in the content of ALT, AST, TG, and TC, and compared with the model group, the inhibitor group had significant reductions in the content of ALT, AST, TG, and TC (all P < 0.05). The model group had significantly higher mRNA and protein expression levels of HDAC1 than the control group, while the inhibitor group had significantly lower expression levels than the model group (all P < 0.05); the model group had significantly lower mRNA and protein expression levels of IRS-1 than the control group, while the inhibitor group had significantly higher expression levels than the model group (all P < 0.05). Conclusion HDAC1 participates in the development and progression of NAFLD by inhibiting the expression of IRS-1 molecule and promoting IR, and the HDAC1 inhibitor pyroxamide can exert a protective effect on the liver by alleviating IR. -
表 1 引物序列
Table 1. Primer sequences
引物 上游(5′-3′) 下游(5′-3′) HDAC1 CTACCGCCCTCACAAAGC ACAGGCCATCGAATACTG IRS-1 TGGATGCAGGTGGATGACT GGCGTGGGTTCTGTTGGT β-Actin GCAGAAGGAGATCACTGCCCT GCTGATCCACATCTGCTGGAA 表 2 不同浓度OA对HepG2细胞活性的影响
Table 2. Effects of different concentrations of oleic acid on the activity of HepG2 cells
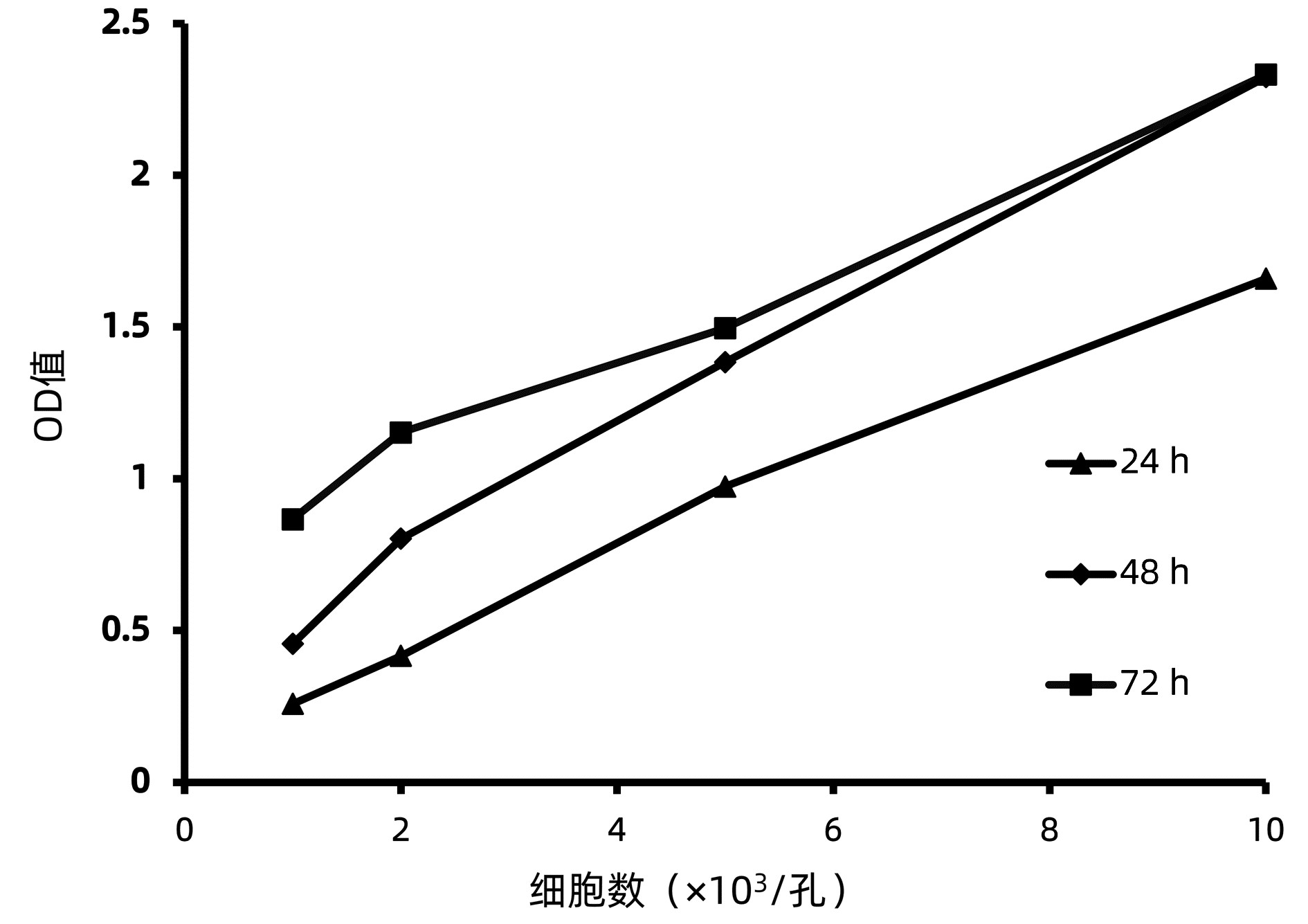
组别 OD值 空白组 0.328±0.008 0 mmol/L组 1.847±0.945 0.125 mmol/L组 1.817±0.251 0.25 mmol/L组 1.653±0.248 0.5 mmol/L组 1.323±0.1561) 1 mmol/L组 1.101±0.0761) 2 mmol/L组 1.028±0.0061) F值 37.395 P值 <0.05 注:与0 mmol/L组比较,1)P<0.05。 表 3 不同浓度Pyroxamide对HepG2细胞增殖活力的影响
Table 3. Effects of different concentrations of Pyroxamide on the proliferation activity of HepG2 cells
组别 OD值 空白组 0.282±0.004 0 μmol/L组 1.390±0.080 5 μmol/L组 1.312±0.145 10 μmol/L组 1.295±0.074 15 μmol/L组 1.278±0.054 20 μmol/L组 1.268±0.164 25 μmol/L组 1.082±0.1361) F值 16.034 P值 <0.05 注:与0 μmol/L组比较,1)P<0.05。 表 4 细胞脂肪变程度比较
Table 4. Comparison of the degree of cellular steatosis
组别 OD值 对照组 0.799±0.045 模型组 1.502±0.1091) 抑制剂组 1.098±0.1082) F值 43.786 P值 <0.05 注:与对照组比较,1)P<0.001;与模型组比较,2)P<0.05。 表 5 各组生化指标比较
Table 5. Comparison of biochemical indicators in each group
组别 ALT(U/L) AST(U/L) TG(mmol/L) TC(mmol/L) 对照组 6.883±0.457 12.937±0.649 0.347±0.083 0.167±0.055 模型组 22.660±1.5011) 51.413±1.5671) 0.657±0.0781) 0.760±0.0701) 抑制剂组 20.577±0.4402) 47.490±1.5402) 0.460±0.0662) 0.570±0.0802) F值 248.987 768.953 12.824 57.644 P值 <0.05 <0.05 <0.05 <0.05 注:与对照组比较,1)P<0.01;与模型组比较,2)P<0.05。 表 6 各组HDAC1、IRS-1 mRNA表达量比较
Table 6. Comparison of HDAC1 and IRS-1 mRNA expression in each group
组别 HDAC1 IRS-1 对照组 0.345±0.005 1.150±0.003 模型组 1.252±0.0061) 0.305±0.0071) 抑制剂组 0.775±0.0062) 0.857±0.0072) F值 19 992.781 16 188.355 P值 <0.01 <0.01 注:与对照组比较,1)P<0.01;与模型组比较,2)P<0.01。 表 7 各组HDAC1、IRS-1蛋白表达量比较
Table 7. Comparison of HDAC1 and IRS-1 protein expression in each group
组别 HDAC1 IRS-1 对照组 0.110±0.015 0.899±0.080 模型组 2.760±0.3201) 0.439±0.0661) 抑制剂组 1.231±0.1502) 0.885±0.0903) F值 127.204 32.636 P值 <0.05 <0.05 注:与对照组比较,1)P<0.01;与模型组比较,2)P<0.01,3)P<0.05。 -
[1] National Workshop on Fatty Liver and Alcoholic Liver Disease, Chinese Society of Hepatology, Chinese Medical Association; Fatty Liver Expert Committee, Chinese Medical Doctor Association. Guidelines of prevention and treatment for nonalcoholic fatty liver disease: A 2018 update[J]. J Clin Hepatol, 2018, 34(5): 947-957. DOI: 10.3969/j.issn.1001-5256.2018.05.007.中华医学会肝病学分会脂肪肝和酒精性肝病学组, 中国医师协会脂肪性肝病专家委员会. 非酒精性脂肪性肝病防治指南(2018年更新版)[J]. 临床肝胆病杂志, 2018, 34(5): 947-957. DOI: 10.3969/j.issn.1001-5256.2018.05.007. [2] WANG YH, GAO Y. Research progress in diagnosis and treatment of non-alcoholic fatty liver disease combinated with type 2 diabetes mellitus[J]. J Jilin Univ(Med Edit), 2020, 286(6): 1324-1331. DOI: 10.13481/j.1671-587x.20200634.王雨涵, 高影. 非酒精性脂肪性肝病并发2型糖尿病诊断和治疗的研究进展[J]. 吉林大学学报(医学版), 2020, 286(6): 1324-1331. DOI: 10.13481/j.1671-587x.20200634. [3] DAY CP, JAMES OF. Steatohepatitis: a tale of two "hits"?[J]. Gastroenterology, 1998, 114(4): 842-845. DOI: 10.1016/s0016-5085(98)70599-2. [4] YANG ZB, PENG H, MA SW. Progress in epigenetics of liver diseases[J]. Chin Hepatol, 2017, 22(5): 381-384. DOI: 10.3969/j.issn.1008-1704.2017.05.002.杨智彬, 彭惠, 马世武. 肝脏疾病的表观遗传学研究进展[J]. 肝脏, 2017, 22(5): 381-384. DOI: 10.3969/j.issn.1008-1704.2017.05.002. [5] CASTELLANO D, RAMOS B, OLIVA W, et al. Genome profiling of H3k4me3 histone modification in human adipose tissue during obesity and insulin resistance[J]. Biomedicines, 2021, 9(10): 1363. DOI: 10.3390/biomedicines9101363. [6] FU S, YU M, TAN Y, et al. Role of histone deacetylase on nonalcoholic fatty liver disease[J]. Expert Rev Gastroenterol Hepatol, 2021, 15(4): 353-361. DOI: 10.1080/17474124.2021.1854089. [7] SODUM N, KUMAR G, BOJJA SL, et al. Epigenetics in NAFLD/NASH: Targets and therapy[J]. Pharmacol Res, 2021, 167: 105484. DOI: 10.1016/j.phrs.2021.105484. [8] GUO YW, TAO L, LIN XY, et al. Role of histone deacetylase 1 in course of mice NAFLD[J]. J Sun Yat-Sen Univ(Medical Sciences), 2016, 37(6): 840-844. https://www.cnki.com.cn/Article/CJFDTOTAL-ZSYK201606008.htm郭云蔚, 陶力, 林显艺, 等. 组蛋白去乙酰化酶1在小鼠非酒精性脂肪肝中的表达和作用[J]. 中山大学学报(医学科学版), 2016, 37(6): 840-844. https://www.cnki.com.cn/Article/CJFDTOTAL-ZSYK201606008.htm [9] RAJAN PK, UDOH UA, SANABRIA JD, et al. The role of histone acetylation-/methylation-mediated apoptotic gene regulation in hepatocellular carcinoma[J]. Int J Mol Sci, 2020, 21(23): 8894. DOI: 10.3390/ijms21238894. [10] JAMES DE, STÖCKLI J, BIRNBAUM MJ. The aetiology and molecular landscape of insulin resistance[J]. Nat Rev Mol Cell Biol, 2021, 22(11): 751-771. DOI: 10.1038/s41580-021-00390-6. [11] YANG Q, VIJAYAKUMAR A, KAHN BB. Metabolites as regulators of insulin sensitivity and metabolism[J]. Nat Rev Mol Cell Biol, 2018, 19(10): 654-672. DOI: 10.1038/s41580-018-0044-8. [12] MAUDE H, SANCHEZ-CABANILLAS C, CEBOLA I. Epigenetics of hepatic insulin resistance[J]. Front Endocrinol (Lausanne), 2021, 12: 681356. DOI: 10.3389/fendo.2021.681356. [13] KHAN S, KOMARYA SK, JENA G. Phenylbutyrate and β-cell function: contribution of histone deacetylases and ER stress inhibition[J]. Epigenomics, 2017, 9(5): 711-720. DOI: 10.2217/epi-2016-0160. [14] KHAN S, KUMAR S, JENA G. Valproic acid reduces insulin-resistance, fat deposition and FOXO1-mediated gluconeogenesis in type-2 diabetic rat[J]. Biochimie, 2016, 125: 42-52. DOI: 10.1016/j.biochi.2016.02.014. -


 PDF下载 ( 2418 KB)
PDF下载 ( 2418 KB)

 下载:
下载: